临沂市食源性单增李斯特菌分子特征分析
作者: 高远 李涛 张晓妮
摘 要:目的:基于全基因组测序分析临沂市食源性单增李斯特菌的毒力基因、耐药基因携带情况及分子分型特征。方法:利用测序数据对27株食源性单增李斯特菌株进行多位点序列分型、毒力基因和耐药基因分析,并构建基于cg-SNP的系统发育树。结果:27株分离菌株共分为10个ST型,其中ST121为优势型别。27株单增李斯特菌有26株菌携带磷霉素(FosX)耐药基因,占96.3%,2株菌携带四环素类(tetM)和甲氧苄氨嘧啶类(dfrG)2种耐药基因,仅有1株菌不耐药。27株菌出现两种毒力缺失,第一种是毒力岛LIPI-1缺失,缺失率为7.4%。第二种是毒力岛LIPI-2中inlF单一缺失,缺失率为40.7%。系统发育树分析显示,27株菌株距离较近,聚集于3个分支中。绝大部分菌株分离自肉制品,占74.1%(20/27)。结论:食源性单增李斯特菌存在多种耐药基因,毒力基因分布以毒力岛(LIPI-1、LIPI-2)为主,具有潜在的致病性。建议临沂市单增李斯特菌专项监测工作在牲畜肉制品的基础上,应加大对冷冻饮品及其乳制品原料、生禽肉类、水果蔬菜类样本的监管,进一步降低单增李斯特菌所致食源性疾病流行风险。
关键词:全基因组测序;单增李斯特菌;基因分型;毒力基因;耐药基因
Molecular Characteristics Analysis of Foodborne Listeria monocytogenes in Linyi City
GAO Yuan, LI Tao, ZHANG Xiaoni*
(Linyi Center for Disease Control and Prevention, Linyi 276000, China)
Abstract: Objective: Based on whole genome sequencing, the virulence genes, drug resistance gene carriers and molecular typing characteristics of foodborne Listeria monocytogenes in Linyi city were analyzed. Method: Using sequencing data, 27 foodborne single-increase Listeria monocytogenes were analyzed by multi-locus sequence typing, virulence genes and drug resistance genes, and a phylogenetic tree based on cg-SNP was constructed. Result: The 27 isolated strains were divided into 10 ST types, among which ST121 was the dominant type. Among the 27 strains of Listeria monocytogenes, 26 strains carried fosfomycin (FosX) resistance genes, accounting for 96.3%, and two strains carried tetracyclines (tetM) and trimethoprim (dfrG) resistance genes, only one strain was not resistant. Two types of virulence were lost in 27 strains, the first was the virulence island LIPI-1 deletion, with a deletion rate of 7.4%. The second was a single deletion of inlF in the virulence island LIPI-2, with a deletion rate of 40.7%. Phylogenetic tree analysis showed that 27 strains were close together and clustered in 3 branches. The vast majority of the strains were isolated from meat products, accounting for 74.1% (20/27). Conclusion: There are a variety of drug resistance genes in foodborne Listeria monocytogenes, and the distribution of virulence genes is dominated by virulence islands (LIPI-1 and LIPI-2), which has potential pathogenicity. It is suggested that the special monitoring work of Listeria monocytogenes in Linyi city should be based on livestock meat products, and should increase the supervision of frozen beverages and their dairy raw materials, raw poultry meat, fruits and vegetables samples, further reducing the risk of foodborne diseases caused by Listeria monocytogenes.
Keywords: whole genome sequencing; Listeria monocytogenes; genotyping; virulence gene; resistance gene
单增李斯特菌是一种引起人类李斯特菌病的常见食源性致病菌,相关文献表明单增李斯特菌感染通常与受污染的食物有关[1],并广泛存在于多种食物中,如生肉、即食食物、牛奶、蔬菜、冰淇淋和水产品等[2]。李斯特菌病临床表现包括败血症和中枢神经系统感染,妊娠相关李斯特菌病可导致早产、流产或死产[3]。美国先前一项研究报告了147例人类李斯特菌病感染病例和33例死亡病例[4]。2015年欧盟共报告了2 224例侵袭性李斯特菌病病例,总病例死亡率为18.8%[5]。在我国,李斯特菌病是一种罕见疾病,尚未被列为法定申报疾病[6]。到目前为止,我国没有关于人群中爆发李斯特菌病的报告。本研究基于全基因组测序技术对27株食源性单增李斯特菌进行耐药基因、毒力基因及分子分型特征分析,为单增李斯特菌病的预防控制提供参考依据。
1 材料与方法
1.1 菌株来源
27株单增李斯特菌分离自临沂市食品安全风险监测样品。
1.2 试剂与仪器
LB1增菌液;李斯特菌显色培养基(法国科玛嘉);脑心浸液琼脂培养基;全基因组DNA提取试剂盒(德国Qiagen公司)。
VITEK2全自动微生物鉴定仪(法国梅里埃公司);CLC Genomics Workbench 21分析软件(德国Qiagen公司)。
1.3 菌株复苏培养
取甘油冻存管保存的27株单增李斯特菌分析用LB1增菌液复苏,接种李斯特菌显色培养基,挑取典型菌落进行纯培养及生化鉴定。
1.4 全基因组测序分析
1.4.1 全基因组测序
依照全基因组DNA提取试剂盒说明书完成DNA的提取,送至诺禾致源科技服务有限公司进行全基因组测序(Whole Genome Sequencing,WGS),将clean data导入CLC Genomics Workbench 21进行组装和分析。
1.4.2 耐药基因、毒力基因及毒力岛分析
通过CLC Genomics Workbench 21分析软件对27株菌进行耐药基因和毒力基因预测。根据毒力基因注释结果对单增李斯特菌毒力岛(Listeria Pathogenicity Island,LIPI)Ⅰ、Ⅱ进行比对分析。
1.4.3 多位点序列分型、谱系分析
基于全基因组测序结果,利用BIGSdb-单增李斯特菌数据库(http://bigsdb.pasteur.fr/listeria/listeria.html)分析基于27株单增李斯特菌菌株的多位点序列分型(Multilocus Sequence Typing,MLST)、谱系及克隆群(Clonal Complex,CC)型。
1.4.4 系统发育树构建
利用在线分析工具CSI Phylogeny 1.4分析27株单增李斯特菌的全基因组序列,构建基于cg-SNP的系统发育树,用iTOL绘制系统发育树[7]。
2 结果与分析
2.1 菌株基本信息
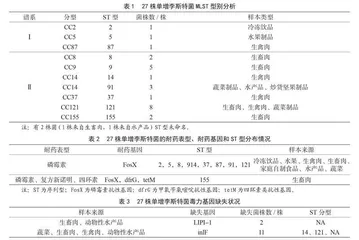
从27株单增李斯特菌来源情况来看,59.3%来自生畜肉(16/27),14.8%来自生禽肉(4/27),其余7株菌分别来自水果制品、冷冻饮品、水产品、炒货坚果制品及家庭自制蔬菜制品(水煮花生、腌酱菜)。27株菌株分为10个ST型,2株菌ST型未命名(NA),ST121为优势ST型,所占比例为29.63%(8/27),其余19株ST型为ST9(5株)、ST91(3株)、ST155(2株)、ST8(2株)等,ST型ST14、ST87、ST2、ST5、ST37仅包含1株分离菌。27株实验菌株分属于2个谱系(Ⅰ和Ⅱ),以谱系Ⅱ为主,共22株(81.5%);分为6个CC型,其中CC9、CC14和CC121为优势CC型,共占63%(17株);分为7个ST型,CC14包括ST14和ST91,其余CC型仅包含1种ST型。详见表1。
2.2 耐药基因分析
耐药基因分析结果显示,27株单增李斯特菌有26株菌携带磷霉素FosX耐药基因,占96.3%。菌株SD22LY0009LM和SDLY20Lmo003均携带四环素类tetM和甲氧苄氨嘧啶类dfrG两种耐药基因,这两株菌都是ST155型,从生畜肉中检出。仅有1株菌SD22LY0024LM不携带耐药基因,详见表2。
2.3 毒力基因分析
27株单增李斯特菌分别检出70~80个毒力基因,根据VFanalyzer毒力基因编码蛋白的注释,选择其中40个毒力基因进行分析。27株单增李斯特菌毒力基因主要集中在毒力岛LIPI-Ⅰ和LIPI-Ⅱ,另外还包括ami、lap基因家族等。27株菌出现两种毒力缺失(见表3),第1种是毒力岛LIPI-Ⅰ缺失,菌株SD22LY0002LM和菌株SSD22LY0024LM出现LIPI-Ⅰ毒力岛上的全部基因缺失,缺失率为7.4%;第2种是毒力岛LIPI-Ⅱ中inlF单一缺失,缺失率为40.7%,其中菌株SSD22LY0024LM又出现了inlF的缺失。27株分离株均未检测到LIPI-Ⅲ和LIPI-Ⅳ毒力岛。与黏附侵袭相关毒力因子ami、iap、lap、lapB、lpeA、pdgA、vip,生存耐受因子bsh、clpC,协助侵入感染因子aut、gtcA、hpt、lplA1、prsA2在27株分离株中均有检出。
2.4 进化树分析
以NC_003210.1为参考菌株,对27株单增李斯特菌进行cg-SNP分析,并据此构建系统发育树,见图1。27株菌株距离较近,聚集于3个分支中。绝大部分单增李斯特菌分离自肉制品,占74.1%(20/27),3个分支中均有分布,主要聚集于C分支中。有2株分离自水产品,2株分离自家庭自制蔬菜制品(水煮花生、腌酱菜)以及1株分离自炒货及坚果制品,主要聚集于分支C中。其余2株分离自冷冻饮品和水果制品的菌株主要聚集于分支A中。27株单增李斯特菌分属10种MLST型,每型包括1~6株菌。分析结果显示,ST型相同的食品同源性较高。ST121为优势ST型,所有ST121菌株具有在碱性和氧化应激条件下生存的能力,提示这部分菌株可在食品生产加工和流通环境中长期存活,增加污染食品和致病的风险[8]。此外ST分型中的ST155、ST87和ST8多为可致病性的ST型。