基于全长转录组测序的金花茶NAC基因家族鉴定及分析
作者: 刘合霞 徐飞凤 杨卓玲 吴晓敏 徐赛颖 李博
摘要 利用生物信息学技术对金花茶(Camellia nitidissima)的全长转录组数据进行筛选,获得了35个金花茶NAC转录因子,将其分别命名为CnNAC1~CnNAC35;随后分析了蛋白理化性质、保守结构域以及系统发育关系;利用RNA-seq数据,对金花茶NAC转录因子在根、茎、叶、花等组织中的表达谱进行了研究;并对金花茶不同开花时期的花瓣表达量与关键类黄酮化合物含量进行Pearson相关性分析。结果表明:金花茶NAC基因家族成员编码的蛋白主要为亲水性的不稳定蛋白,大多定位于细胞核或叶绿体中,多数蛋白都含有NAC基因家族所特有的5个亚保守结构域;系统进化分析中,35个金花茶NAC蛋白被分为 16个亚家族,而NAC2亚家族所包含的蛋白数量最多;金花茶NAC转录因子在不同组织中的表达量存在差异,其中8个金花茶NAC转录因子在花瓣中的表达量相对较高,且与槲皮素、槲皮素-7-O-葡糖苷的变化量显著相关。
关键词 金花茶;类黄酮合成调控;NAC基因家族;进化分析;表达分析
中图分类号 S 685.14 文献标识码 A
文章编号 0517-6611(2025)02-0097-07
doi:10.3969/j.issn.0517-6611.2025.02.022
开放科学(资源服务)标识码(OSID):
Identification and Analysis of NAC Gene Family in Camellia nitidissima Based on Full-length Transcriptome Sequencing
LIU He-xia XU Fei-feng2,YANG Zhuo-ling2 et al
(1.College of Smart Agriculture,Yulin Normal University,Yulin,Guangxi 537000;2.College of Biology and Pharmacy,Yulin Normal University,Yulin,Guangxi 537000)
Abstract In this study,bioinformatics technology was used to screen the full-length transcriptome sequencing data of Camellia nitidissima,and 35 CnNAC transcription factors were obtained,which were named CnNAC1-CnNAC35 respectively.The physicochemical properties,conserved domains and phylogenetic relationships of CnNAC proteins were analyzed.Based on RNA-seq data,the expression profiles of CnNAC transcription factors in root,stem,leaf and flower of Camellia nitidissima were studied.Subsequently,Pearson correlation analysis was conducted between the expression data of Camellia nitidissima petals and the contents of key flavonoids in different flowering periods.The results showed that the CnNAC proteins were mainly hydrophilic and unstable proteins which were mainly located in the nucleus or chloroplast,and most of the proteins contained 5 subconserved domains which was special structure in the NAC gene family.In phylogenetic analysis,35 CnNAC proteins were divided into 16 subfamilies,and NAC2 subfamily contained the most proteins.There were differences in the expression levels of CnNAC transcription factors in different tissues,and the expression levels of 8 CnNAC transcription factors in petals were relatively high which were significantly correlated with the changes of quercetin and quercetin 3-O-rhamnoside-7-O-glucoside.
Key words Camellia nitidissima;Regulation of flavonoid synthesis;NAC gene family;Phylogenetic analysis;Expression analysis
金花茶(Camellia nitidissima)是山茶科山茶属植物,其叶片及花朵中含有皂苷类、多糖、类黄酮等次生代谢物[1-2],可用于治疗高血压、痢疾、咽喉炎和便血等疾病[3]。金花茶中富含的类黄酮化合物不仅是花色形成的关键物质[4-5],还是其具有药用价值的活性成分。近年来,金花茶中与类黄酮合成相关的结构基因及少数MYB转录因子已被克隆[6-8],部分基因的功能已被验证[6,8],金花茶类黄酮合成机理正在逐步阐明,但是bHLH、NAC、WD40等其他类别的转录因子对金花茶中类黄酮合成的调控机制仍知之甚少。
NAC(NAM,ATAF1/ATAF2和CUC2)基因家族是植物中发现的最大转录因子家族之一[9-10],它们在N端具有高度保守的NAC结构域,该结构域由约150个氨基酸残基组成,主要包含5个保守区域(A~E)[11-12];C端区域富含简单的氨基酸重复序列及丝氨酸、苏氨酸、脯氨酸、谷氨酰胺、天冬氨酸、谷氨酸等氨基酸残基,是一个高度分化的转录调控区,具有转录激活或遏制蛋白活性的作用[13-15]。NAC蛋白可与自身或其他NAC蛋白相互作用,形成同源二聚体或异源二聚体,它们互作的区域主要定位于N端的保守NAC结构域[16-17];而通过蛋白互作形成的功能性NAC蛋白二聚体,其表面一侧富含正电荷,该区域可能与DNA结合有关[18-19]。NAC转录因子具有广泛的生物学功能,在植物生长发育、次生代谢物质合成、激素应答、逆境胁迫等方面发挥着重要作用[20-21]。例如,MdNAC029转录因子可激活MdMYB1转录,促进花青素在苹果(Malus pumila)愈伤组织中合成[22];MdNAC52转录因子转录水平在果实着色期间增加,过表达MdNAC52能够促进苹果愈伤组织花色苷的积累[23]。在金花茶不同开花时期的花瓣转录组测序研究中[24-25],发现部分金花茶NAC转录因子在金花茶开花过程中差异表达,它们可能具有调控金花茶中类黄酮合成的功能。目前,基于植物基因组学和生物信息学的手段,植物NAC转录因子基因家族的研究日益增多[26-27],但关于金花茶NAC基因家族以及NAC基因功能的研究尚鲜见报道,因此笔者基于全长转录组测序数据,利用生物信息技术鉴定金花茶NAC基因家族,对其蛋白结构特征进行预测,分析金花茶NAC基因家族成员在不同组织中的表达特征,并对它们的表达量与关键类黄酮化合物的含量进行相关性分析,以期筛选出在金花茶中具有调控类黄酮合成的NAC转录因子。该研究结果将为进一步分析金花茶NAC基因功能及其在类黄酮合成中的作用机制提供科学依据。
1 材料与方法
1.1 材料
在PlantTFDB数据库中(http://planttfdb.gao-lab.org/tf.php?sp=Ath&did=AT5G62610.1)下载拟南芥NAC基因家族的蛋白编码序列,然后将其作为搜寻序列(Query sequence),以金花茶的茎、叶以及不同开花时期的花朵等组织的全长转录组测序数据(Accession number: SRX18217210)作为目标序列,搜索金花茶全长转录组中所包含的NAC基因家族。
1.2 方法
1.2.1 金花茶NAC基因家族成员鉴定。
本地Blast搜索金花茶全长转录组中的NAC基因家族,阈值 E<10-5,用在线软件NCBI (http://www.ncbi.nlm.nih.gov/Structure/cdd/)确定候选基因编码的蛋白序列是否含有NAC蛋白保守结构域,排除不符合要求的基因。
1.2.2 金花茶NAC蛋白质的理化性质、保守结构域以及保守结构元件分析。
通过在线网站ExPASy(http://web.expasy.org/prot-param/)完成对金花茶NAC基因家族成员蛋白质的性质的分析,如等电点、脂肪系数、不稳定系数、平均亲水系数、分子量等;同时利用在线网站WOLF PSORT(https://wolfpsort.hgc.jp/)对蛋白质进行亚细胞定位预测;利用在线工具MEME(http://meme-suite.org/)预测金花茶NAC基因家族成员的保守基序,其中Motifs数量设定为10个,保守位点宽度为6~50,其余参数不变;通过TBtools软件绘制展示结果。对于金花茶NAC蛋白保守结构域的分析,则利用DNAMAN 9.0软件对金花茶NAC蛋白进行多序列比对,分析它们保守结构域的完整性。
1.2.3 金花茶NAC基因家族系统进化树的构建。
采用Neighbor-joining方法,构建金花茶NAC基因家族与拟南芥NAC基因家族的系统进化树,该过程主要利用MEGA10软件完成,其中Boot-strap值设为1 000,而其他参数设为默认值。
1.2.4 金花茶NAC基因家族表达模式分析及相关性分析。
利用金花茶根、茎、叶、不同开花时期的花瓣、不同发育时期花芽的转录组测序数据,分析金花茶NAC基因家族在不同组织中的表达量,然后使用R语言软件中的Heatmap工具绘制表达量热图;并将金花茶不同开花时期的花瓣表达量与槲皮素(Quercetin)、山奈酚(Kaempferol)、槲皮素-7-O-葡糖苷(Quercetin-3-O-rhamnoside-7-O-glucoside,Qu7G)、槲皮素-3-O-葡糖苷(Quercetin-3-O-glucoside,Qu3G)等类黄酮化合物的含量进行Pearson相关性分析,研究NAC基因的表达量与关键类黄酮化合物之间的相关性。
2 结果与分析
2.1 金花茶NAC基因家族的筛选及蛋白特性预测
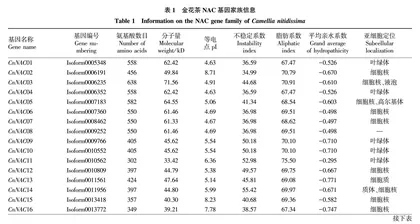
利用生物信息学技术手段,在金花茶全长转录组中筛选获得35个有完整ORF的NAC基因家族成员,分别将其命名为CnNAC1~CnNAC35(表1);其中CnNAC03的氨基酸序列最长,有638个氨基酸残基,CnNAC35的氨基酸序列最短,仅有292个氨基酸残基。对35个金花茶NAC蛋白的物理特性进行预测,发现它们的分子量分布范围为33.30~71.56 kD,等电点(isoelectric point,pI)的分布范围在4.62~8.98,其中26个为酸性蛋白,9个为碱性蛋白;不稳定系数为33.31~54.42,其中17个属于稳定蛋白,18个属于不稳定蛋白;而金花茶NAC蛋白的平均亲水指数分布范围为-0.892~-0.295,结果表明所有金花茶CnNAC蛋白均为亲水性蛋白;它们的脂肪系数为55.65~75.50。对35个金花茶NAC蛋白进行亚细胞定位预测,显示这些蛋白主要分布在细胞核和叶绿体中,少数定位在液泡、质体和高尔基体中。