石斛酚对小鼠黑色素瘤细胞转录组影响的测序比对分析
作者: 阿卜杜米吉提•艾海提 韩雪 戴小华 史慧君 姚刚 向志宇
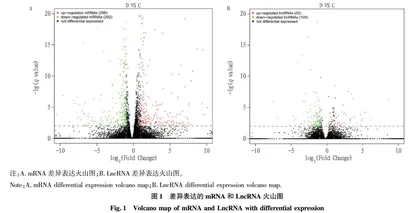
摘要 为了鉴定和分析石斛酚对黑色素瘤细胞作用过程中差异表达的mRNA和LncRNA,以小鼠黑色素瘤细胞为试验对象,采用RNA-Seq技术和生物信息学方法对石斛酚组(160 μmol/L)和对照组(0 μmol/L)进行转录组测序分析,运用DESeq软件鉴定2组间差异表达的mRNA和LncRNA,对差异表达的mRNA进行GO功能注释和KEGG信号通路富集分析,同时对差异表达的LncRNA靶基因进行预测分析。结果显示,2组间存在588个差异表达的mRNA。与对照组相比,石斛酚组显著上调表达296个mRNA和显著下调表达292个mRNA;588个差异表达基因共富集在66个GO功能条目上,主要包括生物学过程、细胞组分和分子功能;富集的KEGG信号通路主要包括P53信号通路、雌激素信号通路、范可尼贫血信号通路、MAPK信号通路和磷脂酰肌醇信号通路等。另外,2组间存在161个差异表达的LncRNA。与对照组相比,石斛酚组显著上调表达52个LncRNA和显著下调表达109个LncRNA;通过对差异表达的LncRNA靶基因进行预测分析,共获得259个顺式作用的靶基因和6 950个反式作用的靶基因。由此可见,石斛酚处理可以改变黑色素瘤中多种mRNA和LncRNA的表达,这些差异表达的mRNA和LncRNA可能是石斛酚治疗黑色素瘤的药物靶点。
关键词 黑色素瘤;石斛酚;转录组测序
中图分类号 S852.21 文献标识码 A 文章编号 0517-6611(2025)03-0092-04
doi:10.3969/j.issn.0517-6611.2025.03.018
开放科学(资源服务)标识码(OSID):
Sequencing Comparative Analysis of the Effects of Gigantol on the Transcriptome of Mouse Melanoma Cells
Abudumijit·Ahat, HAN Xue, DAI Xiao hua et al
(College of Veterinary Medicine, Xinjiang Agricultural University, Urumqi, Xinjiang 830052)
Abstract In order to identify and analyze the differentially expressed mRNA and LncRNA in the process of dendrophenolics acting on melanoma cells in mouse, the transcriptome sequencing of gigantol group (160 μmol/L) and control group (0 μmol/L) was carried out by RNA Seq technology and bioinformatics methods. The differentially expressed mRNA and LncRNA were identified by DESeq software. The differentially expressed mRNA was annotated by GO function and enriched by KEGG signal pathway, and the target gene of differentially expressed LncRNA was predicted and analyzed.The results showed that there were 588 differentially expressed mRNA between the two groups. Compared with the control group, 296 mRNA were significantly up regulated and 292 mRNA were significantly down regulated in the gigantol group. A total of 588 differentially expressed genes were enriched in 66 GO functional items, mainly including biological processes, cellular components and molecular functions. The enriched KEGG signaling pathways mainly included P53 signaling pathway, estrogen signaling pathway, Fanconi anemia signaling pathway, MAPK signal pathway and phosphatidylinositol signal pathway. In addition, there were 161 differentially expressed LncRNAs between the two groups. Compared with the control group, 52 LncRNAs were significantly up regulatedand 109 LncRNAs were significantly down regulated in the gigantol group.The prediction and analysis of differentially expressed LncRNA target genes showed that 259 cis acting target genes and 6950 trans acting target genes were obtained. In conclusion, gigantol treatment can change the expression of various mRNA and LncRNA in melanoma, and these differentially expressed mRNAs and LncRNAs may be the drug targets of gigantol in the treatment of melanoma.
Key words Melanoma;Gigantol;Transcriptome sequencing
基金项目 新疆维吾尔自治区自然科学基金(青年基金)项目(2019 D01B24);新疆农业大学前期资助项目(XJAU201707)。
作者简介 阿卜杜米吉提·艾海提(1997—),男,新疆库车人,硕士研究生,研究方向:基础兽医学。
*通信作者,讲师,硕士,从事基础兽医学研究。
收稿日期 2023-08-13
黑色素瘤是一种由于黑色素细胞增殖无法受到控制而引起的最具侵袭性肿瘤,主要发生于人或动物皮肤的无毛部位。虽然皮肤黑色素瘤占所有皮肤恶性肿瘤的比例不到5%,但由于其具有较高的侵袭性和转移性[1],死亡率在皮肤恶性肿瘤中排第一名,严重危害着人和马属动物的健康。目前治疗黑色素瘤的药物只有少数几种,如达卡巴嗪、替莫唑胺等,但治疗有效率较低。因此,研发新型有效、低毒的抗黑色素瘤药物对于临床治疗黑色素瘤患者和动物具有重要意义。
中医药在抗肿瘤和抗病毒等方面历史悠久,从中草药提取物中筛选低毒、安全、有效的成分作为抗肿瘤药物一直是学界的研究热点[2]。现代药理研究表明,石斛中酚酸类化合物具有抗肿瘤、抗衰老、增强人体免疫力、抗凝血及扩张血管等活性[3-6],而石斛酚对非小细胞肺癌、乳腺癌、结肠癌和肝癌等[7-9]具有显著的抗癌活性。然而,目前石斛酚对黑色素瘤的作用机制尚未见报道。LncRNA与mRNA在恶性黑色素瘤进化发展中扮演着至关重要的角色。笔者探讨了石斛酚对黑色素瘤中LncRNA和mRNA表达的影响,以期为将石斛酚应用于黑色素瘤的防治提供理论基础。
1 材料与方法
1.1 主要试剂
供试石斛酚购自上海纯优生物科技有限公司;小鼠黑色素瘤细胞系B16购自中国科学院上海细胞库;DMSO购自福州奥研实验器材有限责任公司;DMEM高糖培养基、0.25% Trypsin & 0.02%EDTA、青霉素-链霉素溶液、细胞培养皿(60 mm)均购自北京索莱宝科技有限公司;胎牛血清FBS购自杭州四季青生物工程材料有限公司。
1.2 试验药物与细胞
1.2.1
小鼠黑色素瘤细胞系B16。小鼠黑色素瘤细胞系B16用含10% FBS、100 U/mL青霉素和100 mg/mL链霉素的DMEM高糖培养基培养,在37 ℃、5% CO2条件下置于恒温培养箱中培养。每1~2 d更换1次新鲜培养基。一旦细胞融合达到80%~90%,就将细胞传代培养,并将第3代细胞整合用于后面的试验。
1.2.2
石斛酚。石斛酚使用0.004%的DMSO进行溶解,使其浓度达到44 mmol/L,然后用DMEM高糖培养基稀释成相应的浓度,4 ℃下备用。
1.3 试验分组及药物干预
将小鼠黑色素瘤细胞系B16分成2组,即对照组和石斛酚组,依次加入0、160 μmol/L石斛酚处理24 h后收集细胞,用于RNA-Seq试验。
1.4 RNA-Seq文库构建和测序数据预处理
提取对照组和石斛酚组的RNA,由生工生物工程(上海)股份有限公司进行质检,质检合格后建立全转录组文库。2组样本经PCR扩增及纯化、文库质检后,上机进行全转录组测序。随后,将获得的原始数据用Trimmomatic软件进行过滤,采用滑窗法去除含有带接头、低质量的序列,并根据mRNA、LncRNA的特点,保证获得Clean数据;从Clean数据中抽取10 000条序列利用NCBINT数据库进行blastn比对,取e值≤10-10且相似度>90%、覆盖度>80%的比对结果,计算其物种分布。
1.5 DE mRNAs和DE LncRNAs的筛选与分析
针对对照组和石斛酚组的Clean数据,利用R语言中的vegan package进行主成分分析(principal component analysis,PCA)和样本间相似程度分析,然后使用DESeq2软件进行差异分析,筛选条件设置如下:P值≤0.05且差异倍数|Fold Change|≥2,获得2组间显著差异表达的mRNA和LncRNA。最后,利用GO、KEGG数据库对差异表达的mRNA进行GO功能和KECG通路富集分析。
1.6 LncRNA靶基因预测及功能注释
利用Bedtools算法预测LncRNA靶基因,预测LncRNA靶基因过程中所使用的阈值参数为S≥150,ΔG≤-125.58 kJ/mol和Demand strict 5′seed pairing,其中S是指匹配区域内single residue pair match scores;ΔG为双链形成时所需的自由能。
2 结果与分析
2.1 石斛酚组和对照组测序数据质量分析
通过建库测序,从对照组中分别获得9 271 408 463、6 950 508 120、7 374 448 953个有效总碱基数,从石斛酚组中分别获得7 779 118 532、9 921 516 979、11 140 731 042个有效总碱基数;6组样本的GC含量为53.16%~55.18%,碱基质量Q30数目分别为8 621 895 070、6 489 612 063、6 870 254 215、7 229 617 505、9 194 455 091、10 335 601 705,碱基质量值Q30在92.67%以上,可见样本质量符合测序要求。