野生及人工饲养牟氏螺蛳肠道微生物多样性的比较研究
作者: 郑健 杨锁柱 周艳萍 向泓铨 赵津
摘要 通过3代测序技术调查并比较野生和人工饲养的牟氏螺蛳肠道菌群组成和多样性差异,并分析了造成这些差异的原因。结果表明,Proteobacteria、Firmicutes和Bacteroidetes是野生和人工饲养牟氏螺蛳肠道微生物的主要门水平组成。人工饲养和野生牟氏螺蛳的肠道微生物存在显著差异,这一差异是由于在人工饲养驯化过程中低丰度物种的丢失造成的。该研究首次揭示了牟氏螺蛳肠道微生物多样性及其在人工饲养过程中肠道微生物群落构建机制的差异,为牟氏螺蛳的研究和保护提供了一定的理论基础。
关键词 牟氏螺蛳;肠道菌群;多样性;实时单分子测序
中图分类号 S 917 文献标识码 A 文章编号 0517-6611(2023)03-0072-04
doi:10.3969/j.issn.0517-6611.2023.03.016
Comparison Study of Intestinal Microbial Diversity between Wild and Cultured Margarya monodi
ZHENG Jian1,2, YANG Suo-zhu1, ZHOU Yan-ping1 et al
(1.Yuxi Agriculture Vocation-Technical College, Yuxi,Yunnan 653100;2.Yuxi-BGI Joint Sequencing Center for Plateau Agriculture, Yuxi,Yunnan 653100)
Abstract Based on the the third generation sequencing technology, the composition and diversity differences of intestinal flora between wild and cultured Margarya monodi were investigated. And the reasons for these differences were analyzed. The results showed that main phylum composition of intestinal flora in wild and cultured M. monodi were Proteobacteria, Firmicutes and Bacteroidetes. There were significant differences of intestinal flora between wild and cultured M. monodi, which were caused by the loss of low-abundance species in the process of artificial breeding and domestication. The differences of intestinal microbial diversity and the construction mechanism of intestinal microbial community in the process of artificial breeding were firstly revealed in this research, which provided some theoretical basis for the study and protection of M. monodi.
Key words Margarya monodi;Intestinal flora;Diversity;Real-time single molecule sequencing
基金项目 云南省教育厅科学研究基金项目(2021J1049)。
作者简介 郑健(1987—),男,云南通海人,助教,硕士,从事微生物资源开发与利用、环境微生物研究。通信作者,讲师,硕士,从事动物病疫方面的研究。
收稿日期 2022-03-10
螺蛳是一种软体动物,隶属田螺科(Viviparidae)螺蛳属(Margarya),仅分布在我国云南省的高原湖泊中,为我国特有物种[1-2]。螺蛳不仅是高原湖泊物种多样性的重要组成部分,而且被列为《国家重点保护动物名录》二级保护动物。除了在物种多样性方面具有重要的生态地位外,螺蛳在重金属污染、水体有机污染等方面也有着独特的生态指示功能[3-4]。滇池是云南省最大的高原淡水湖泊。王丑明等[5]2011年调查结果表明,滇池中的螺蛳属包括螺蛳(Margarya melanioides)、牟氏螺蛳(M. monodi)、光肋螺蛳(M.mansugi)、长螺蛳(M.elongata)、 乳顶螺蛳(M.tropidophora)等。其中,牟氏螺蛳(Margarya monodi)是螺蛳属中的一个特殊物种,仅分布在滇池。20世纪80年代以来水体污染、过度捕捞越来越严重,包含牟氏螺蛳在内的螺蛳种群数量急剧减少,2009年螺蛳被世界自然保护联盟列为极危物种。牟氏螺蛳的种群变化不仅是螺蛳属物种多样性变化的指征,而且是滇池水环境的重要指征。然而,目前对牟氏螺蛳在内的螺蛳属生物的相关研究还相对较少,特别是对螺蛳生存的微生物环境等方面缺乏了解。
近年来,随着测序技术的进步,学者对人和其他动物肠道微生物的研究证实了肠道微生物对宿主健康、行为有着巨大的影响[6-7]。对于水生生物及软体动物,肠道微生物同样与它们的生存环境及健康密切相关[8-10]。肠道菌群作为螺蛳体内微生物生态的一部分,其群落构建的机制及其对螺蛳的影响尚不清楚。笔者通过使用3代全长扩增子测序技术对牟氏螺蛳肠道菌群的16S rRNA基因进行高通量测序,比较野生和人工饲养牟氏螺蛳肠道微生物组成和多样性的差异,旨在揭示螺蛳肠道菌群的形成机制,为螺蛳的保护、驯养提供一定的参考依据。
1 材料与方法
1.1 样品搜集
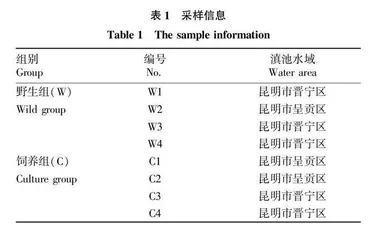
2021年6月,从云南省昆明市的滇池水域采集并鉴定了8个活体牟氏螺蛳。选取其中4个进行解剖,将其肠道内容物洗脱至5 mL无菌水中,-80 ℃下冷冻保存,作为野生组(W组)用于后续测序分析;其余4个牟氏螺蛳放置在水箱中,使用25 ℃滇池水,以蒸煮后的海菜花和南瓜以及市售硅藻粉和螺旋藻粉为食物,在通气条件下喂养2个月,然后进行解剖,作为饲养组(C),取样方法与W组相同。具体样品信息见表1。
1.2 DNA提取及3代全长扩增子测序
含有肠道内容物的液体混匀后,吸取250 μL,使用ZymoBIOMICS DNA Miniprep Kit进行总DNA提取。提取后的DNA使用0.8%琼脂糖凝胶电泳检测DNA完整性,随后使用Tecan F200进行核酸浓度检测(PicoGreen染料法)。质量检测合格的DNA使用引物对8F(5′-AGAGTTTGATCATGGCTCAG-3′)和1492R(5′-CGGTTACCTTGTTACGACTT-3′)进行全长16S rRNA基因扩增。PCR程序如下:预变性98 ℃ 30 s,1个循环;98 ℃变性5 s,54 ℃退火15 s,72 ℃延伸45 s,28个循环;72 ℃ 2 min,1个循环;4 ℃保温。PCR产物使用1% 琼脂糖凝胶进行目的片段电泳检测。检测合格的样品取目的条带使用Zymoclean Gel Recovery Kit(D4008)进行回收,并使用Tecan F200定量;将所有样品的待测产物等摩尔量混合后,使用Nanopore Ligation Sequencing Kit(SQK-LSK109)以及Nanopore Flow Cell Priming Kit(EXP-FLP002)构建测序文库。最后,使用Nanopore GridION Mk1测序仪及MinION Flow Cell R9.4.1芯片,送至成都罗宁生物科技有限公司完成3代测序工作,单个样本测序量保证3万条以上。
1.3 数据分析
原始Fast5序列使用Guppy软件进行碱基解析(base calling),获得Fastq格式的序列文件。Fastq文件使用qcat进行样品拆分。对拆分后的数据进行质量控制,保留序列平均质量 >Q10、长度1 300~1 600 bp的序列。使用Kraken2软件结合SILVA数据库v138对质量控制后的序列进行物种注释,并合并每个物种的序列数目,构建物种丰度表(OTU表)。随机选取每个物种内的一条序列作为代表性序列,使用MUSCLE进行多重比对,随后使用FastTree构建Maximum-likelihood系统发育树。
使用R语言V4.1.0进行数据统计分析。使用vegan包函数diversity计算样品的α多样性指数Chao1和Simpson。α多样性的组间Kruskal-Wallis秩和检验使用kruskal.test函数。使用GUniFrac包分别计算Weighted(丰度加权)UniFrac和Unweighted(非丰度加权)UniFrac β多样性矩阵。NMDS(非度量多维尺度)分析使用vegan包中的metaMDS函数。β多样性矩阵的组间ANOSIM分析使用vegan包中的anosim函数进行。random forest差异物种分析使用random forest包。
2 结果与分析
2.1 野生和饲养牟氏螺蛳的肠道菌群群落组成
从群落组成角度,该研究重点关注了门水平和种水平的群落组成及组间差异。从门水平来看,野生组和饲养组的主要物种组成差异不大(图1)。相对丰度在1%以上的门为Proteobacteria、Firmicutes和Bacteroidetes。从种水平来看,主要使用韦恩图分析了组间独有和共有物种的组成情况。2组间共有物种670个,这些物种占总体物种丰度的96.97%。 W组独有物种为262个、C组独有物种为159个,分别占总体物种丰度的1.93%和1.10%。由此可见,野生和饲养牟氏螺蛳的微生物组成总体区别不大,虽然2组存在多种稀有物种,但占比极低。由此可见,野生牟氏螺蛳在人工饲养驯化过程中,核心肠道菌群保持了相对的鲁棒性。
2.2 野生和饲养牟氏螺蛳的肠道菌群多样性差异
群落多样性分别从α多样性和β多样性2个角度进行了分析。α多样性表征的是样品的多样性指标,图2分别展示了Chao1指数和Simpson指数的多样性结果。总体来看,2个α多样性指数都表现出W组比C组有着更高的多样性。其中Chao1指数W组和C组间存在显著差异(P<0.05)。Simpson指数虽然2组间存在一定差异,但2组间差异不显著。究其原因,Chao1指数对稀有物种较Simpson指数有更多的加权。由此可见,从α多样性角度来看,W组和C组间的差异更多地体现在稀有物种的丢失导致的群落丰富度和均匀度的降低。
组间的多样性差异使用β多样性来表征,这里采用基于UniFrac距离矩阵的β多样性NMDS排序分析(图3)。基于加权的UniFrac距离的NMDS分析(图3A)中,W组和C组并没有出现明显的分隔;基于非加权的UniFrac距离的NMDS分析(图3B)中,W组和C组存在明显的各自成簇趋势。基于2种UniFrac距离矩阵的ANOSIM的定量差异统计分析也证实了这一结果:加权的UniFrac组间ANOSIM分析结果中r=0.281 3、P=0.135,表示不存在显著差异;非加权的UniFrac组间ANOSIM分析结果中r=0.708 3、P=0.031,P<0.05表示存在显著差异。这表明群落构建的组间差异主要是由于物种组成的变化而不是物种丰度的变化造成的。
上述基于多样性的分析结果表明,野生和饲养牟氏螺蛳的肠道微生物存在差异。这一差异是由于稀有菌群在驯化饲养过程中的丢失造成的。由于饲养过程中主要是食物产生了变化,因此推测造成这一结果的原因有2个:一是由于在饲养过程中食物是经过高温灭菌的,所以食物所携带的微生物变少,饲养螺蛳失去了来自食物的微生物在其肠道中的持续定殖,因此肠道微生物呈现多样性降低的过程。二是食物种类的变化对宿主肠道微生物有较大的影响。食物的类型会显著影响宿主肠道微生物的多样性,例如Bolnick等[11]研究发现三刺鱼和河鲈的肠道微生物多样性与食物的多样性呈负相关,而牟氏螺蛳的食物多样性降低带来了肠道菌群多样性的降低。