Cre/loxP系统应用于毕赤酵母基因敲除的研究进展
作者: 王未鲜 韩铭海
摘要 毕赤酵母是重组蛋白最常用的表达系统之一,蛋白高水平表达策略一直是领域内最重要的研究课题之一。在后基因组时代,高效而便捷的基因改造是一种有效提高毕赤酵母蛋白表达能力的手段,其中由Cre/loxP系统介导的基因改造技术受到了科研人员的广泛关注。综述了Cre/loxP系统的基本原理、应用于毕赤酵母基因敲除的技术路线及其关键的技术问题。
关键词 毕赤酵母;Cre/loxP;基因敲除
中图分类号 Q 78 文献标识码 A 文章编号 0517-6611(2023)15-0010-04
doi:10.3969/j.issn.0517-6611.2023.15.003
Research Progress on Gene Knockout in Pichia pastoris Mediated by Cre/loxP System
WANG Wei-xian1, HAN Ming-hai2
(1. Analytical and Testing Center, Guizhou Institute of Technology, Guiyang, Guizhou 550000;2.College of Food and Pharmaceutical Engineering, Guizhou Institute of Technology, Guiyang, Guizhou 550000)
Abstract Pichia pastoris is one of the most commonly used systems for expressing recombinant proteins, and the strategy for high-level protein expression is one of the most important research topics in this field. In the post-genomic era, efficient and convenient genetic modification is an effective means to improve the ability of protein expression in Pichia pastoris. Among them, the gene modification technology mediated by Cre/loxP system has attracted extensive attention of researchers. In this paper, the basic principle of Cre/loxP system, the technical route and the key technical issues of gene knockout in Pichia pastoris are summarized.
Key words Pichia pastoris;Cre/loxP;Gene knockout
毕赤酵母(Pichia pastoris,最新系统命名为Komagataella phaffii)是重组蛋白最常用的表达系统之一[1-2]。相比较于其他表达系统,毕赤酵母具有明显的优势:低廉的培养成本、较高的培养密度、易于操控的启动子、易于基因改造操作、优良的蛋白分泌能力、不分泌热原物质、具有真核细胞的翻译后修饰,尤其是由其表达的多肽都具有蛋白活性[1-2]。然而由此宿主表达的一些有价值的蛋白产品水平仍然较低,因此,促进异源蛋白表达的技术和策略依然是该领域最重要的研究课题之一[3-4]。毕赤酵母的基因组测序已完成,并已公开发布[5-6],在后基因组研究中,以提高毕赤酵母蛋白表达能力为目的的高效而操作便捷的基因改造技术受到科研人员广泛关注。其中,多篇文献报道了科研人员利用Cre/loxP系统成功地完成了毕赤酵母的基因敲除操作[7-12],充分显示了该系统应用于这种酵母基因改造的可靠性。
1 Cre/loxP系统
来自P1噬菌的Cre/loxP系统由Cre重组酶和loxP位点组成,而Cre/loxP介导的基因重组可应用于酵母、拟南芥、棉花、水稻、果蝇、小鼠和斑马鱼等多种真核细胞的基因改造[13-14]。截至2023年2月,以“Cre recombinase”为关键词,通过PubMed搜索引擎可检索到超过7 000篇的文献,这也说明了Cre/loxP系统应用于细胞基因改造技术的广泛性和有效性。
1.1 Cre重组酶
Cre重组酶是由343个氨基酸组成的单体蛋白,分子量约为38 kDa[13-14],其有2个结构域:NTD(N-末端结构域,由110个氨基酸残基组成)和CTD(C-末端结构域,由210个氨基酸残基组成)。Cre重组酶蛋白的活性中心位于CTD结构域中,主要由Arg173-His289-Arg292三联体结构、2个亲核性氨基酸残基Trp315、Try324以及Glu176、Lys201构成,其中Try324残基具有切割活性,另外Arg259的功能与Cre重组酶的催化活性和loxP位点特异性有关[15]。Cre重组酶催化2个loxP位点之间的重组反应,不需要任何宿主辅因子或辅助蛋白[13-14]。
1.2 loxP位点
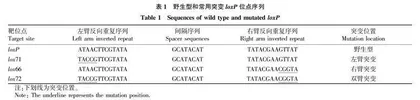
loxP位点是1个长度为34 bp的序列,由2个13 bp的反向重复序列和中间8 bp间隔序列组成,由于8 bp间隔序列是不对称的,这确定了loxP位点的方向[16-17]。在DNA分子链上,如果2个loxP位点方向相反,则在Cre重组酶的作用下,2个loxP位点之间的DNA序列发生倒位,这种情况一般不具有基因改造应用价值;如果2个loxP位点方向相同,则Cre重组酶可以高效地删除它们之间的DNA序列,产生一个新的loxP位点[13-14]。但是,新的loxP位点依然被Cre识别,并参与Cre/loxP介导的重组反应,这不利于细胞的基因改造。为了解决这一问题,研究人员使用了2种突变的loxP位点,即lox71和lox66[13-14],它们在左臂反向重复序列或右臂反向重复序列中含有5 bp的突变(表1),lox71和lox66位点可以被Cre蛋白有效识别,重组反应产生一个新的loxP位点——lox72。lox72是一个惰性的位点,其左臂反向重复序列和右臂反向重复序列中同时含有5 bp的突变,与Cre重组酶的亲和力极微弱,因此不参与Cre/loxP介导的重组反应,从而实现lox71和lox66之间的DNA片段永久删除[13-14,18]。
2 Cre/loxP系统应用于毕赤酵母基因敲除
2.1 基本原件和主要过程
利用Cre/loxP系统敲除基因的主要核心问题是如何构建基因敲除盒(gene disruption cassette)。基因敲除盒主要包含3个基本原件[13-14]:①筛选标记基因,一般常用的是抗生素Zeocin抗性标记[7-9,11-12],有些是G418抗性标记[10];②位于筛选标记基因上下游的2个突变的loxP位点(一般是lox71和lox66);③靶基因上游和下游同源臂序列。利用Cre/loxP系统敲除靶基因的过程主要分为两步[13-14]:①靶基因与基因敲除盒中的同源臂发生同源重组,基因敲除盒插入靶基因序列,破坏靶基因;②Cre重组酶催化lox71和lox66位点发生重组,删除这2个loxP位点之间的DNA序列,切除筛选标记,靶基因序列中留下1个惰性的loxP位点(即lox72),完成靶基因的敲除。
2.2 技术路线和策略
Pan等[7]报道了一种新颖的构建基因敲除盒技术路线:①将Cre蛋白基因克隆到pPICZA质粒上,通过PCR获得Cre-ZeoR序列(ZeoR是Zeocin抗性基因Sh ble的序列);②通过融合PCR技术融合3段DNA序列,将lox71和lox66插入Cre-ZeoR序列两端,构建基因敲除盒U-lox71-Cre-ZeoR-lox66-D(U为靶基因上游同源臂,D为靶基因下游同源臂);③将基因敲除盒电转毕赤酵母,插入靶基因序列中;④甲醇诱导Cre蛋白表达,激活Cre/loxP系统删除lox71与lox66之间的DNA序列。这种方法的优点是可以采用较少的步骤来完成基因敲除盒的构建,但是同时也存在不足:①融合PCR技术对科研人员的操作经验有一定的要求,并且由于引物中存在lox66和lox71序列,其含有较多的重复序列,进一步增加了融合PCR的难度,这可能要通过多次的尝试甚至要更换不同的引物才能够获得成功;②构建的基因敲除盒U-lox71-Cre-ZeoR-lox66-D无法克隆到质粒中进行保存和扩增,因此如果要敲除多个基因,则需要构建多个不同的基因敲除盒序列,这将是一项繁重的实验操作。另外Pan等[7]还推测,多次PCR可能导致基因敲除盒序列发生一定突变,可能会影响Cre/loxP系统的效率。
Han等[8]报道了一种改进的两步法策略(图1),将Cre蛋白基因和loxP序列安置在不同的质粒中,构建的中间序列可以插入质粒中保存和扩增,具体技术路线如下:①从pPICZαA获得ZeoR序列,通过PCR构建lox71-ZeoR-lox66序列原件,并插入克隆质粒中保存;②在获得lox71-ZeoR-lox66序列基础上,克隆靶基因上下游同源臂序列,构建U-lox71-ZeoR-lox66-D基因敲除盒序列;③通过电转将基因敲除盒导入毕赤酵母,嵌入靶基因;④将Cre蛋白基因克隆到pPICZαA质粒中,构建pPICZαA/Cre质粒,并将HIS4基因(源于pAO815质粒)插入pPICZαA/Cre获得辅助质粒pPICZαA/Cre/his4;⑤通过电转,将辅助质粒转化到上述已嵌入基因敲除盒的毕赤酵母中,产生U-lox71-ZeoR-pPICZαA/Cre/his4-lox66-D序列(重组位点在ZeoR序列中);⑥甲醇诱导Cre蛋白表达,删除ZeoR-pPICZαA/Cre/his4序列。两步法技术路线的优点是:①易于操作,可以避免有技术难度的融合PCR操作;②lox71-ZeoR-lox66或U-lox71-ZeoR-lox66-D中间序列都可以克隆并保存在质粒中,有利于多基因敲除操作;③该方法中构建的辅助质粒pPICZαA/Cre/his4还可以直接应用于其他不同基因的敲除。总之,相比Pan等[7]报道的方法,虽然增加了操作步骤(增加了一次毕赤酵母的转化操作),但是对于初次接触分子生物学的研究人员是十分友好的,而且特别适用于多基因敲除的操作。
如果需要敲除的靶基因是毕赤酵母维持正常生长所必需的,那么用传统的基因敲除方法可能就得不到所需的突变菌株。对于这个技术难题,Shibui等[9]提出了一种有效的解决方案:在基因敲除盒U-lox71-Cre-ZeoR-lox66-D序列中插入靶基因对应的救援基因(rescue gene),当基因敲除盒序列破坏靶基因后,基因敲除盒序列中的救援基因就会弥补靶基因的功能,使毕赤酵母细胞的生长表型不会发生改变。
2.3 Cre/loxP系统在毕赤酵母中应用的技术问题
2.3.1 PAOX1驱动Cre蛋白表达。
启动子PAOX1具有较高的转录活性[1-2],是毕赤酵母中最常用异源蛋白表达的启动子之一。大部分的文献报道,应用于毕赤酵母的Cre/loxP系统的Cre蛋白都是利用PAOX1来表达的,这也使得该系统在这种细胞中表现出了较高的活性。Shibui等[9]报道,在30 ℃,甲醇诱导PAOX1表达Cre蛋白6 h后,96%的毕赤酵母细胞完成了Cre/loxP系统介导的重组;Han等[8]报道,在28 ℃,甲醇诱导48 h,100%的毕赤酵母细胞完成了此重组反应。另外,即使lox71与lox66之间有冗长的序列,Cre蛋白依然能高效地完成催化重组。例如,Han等[8]报道,当lox71与lox66之间序列长达10 kbp时,经过48 h的甲醇诱导,基因重组效率依然达到了100%。
在毕赤酵母中,PAOX1是受到严格调控的,在有葡萄糖、乙醇、甘油等碳源存在条件下,PAOX1的转录活性是受到强烈抑制的[1-2];但Shibui等[9]报道,在PAOX1活性被抑制的情况下,毕赤酵母中的Cre/loxP系统还是表现出了微弱活性。
在大肠杆菌中,PAOX1没有受到细胞的调控,可能表现出一定的活性,由PAOX1驱动的Cre蛋白基因出现了一定程度的渗漏表达(leakage expression)[19-20],这就导致Cre/loxP系统表现出了一定的活性。因而,含有lox71-PAOX1-Cre-lox66序列的质粒在大肠杆菌中不稳定[8,19-20],这也许是部分研究者选择通过融合PCR构建基因敲除盒的主要原因。