骆马湖国家级水产种质资源保护区20种鱼类的COI条形码构建和分析
作者: 李大命 刘洋刘燕山 张彤晴 殷稼雯 张伟 穆新武 蒋琦辰
摘要 通过PCR扩增和测序,获得骆马湖国家级水产种质资源保护区20种鱼类的201条线粒体COI条形码,并对20种鱼类的COI条形码进行分析。结果显示,COI基因序列碱基的平均含量分别为A 24.4%、C 28.3%、T 28.5%和G 18.8%,碱基AT含量(52.9%)大于GC含量(47.1%)。20种鱼类的种内和种间遗传距离分别为0~0.017 7和0.008 0~0.308 8,遗传距离平均值分别为0.002 9和0.202 5,种间遗传距离为种内遗传距离的69.8倍。科内属间、目内科间和目间的遗传距离分别为0.150 3、0.206 1和0.236 4,表明遗传距离随着分类界元上升有增加趋势。分子系统学分析结果显示,20种鱼类在系统进化树上均为单系,与形态学分类结果一致。该研究首次构建了骆马湖国家级水产种质资源保护区20种鱼类的COI条形码,可为保护区渔业资源管理及生物多样性保护提供科学依据。
关键词 DNA条形码;COI基因;遗传距离;物种鉴定;骆马湖
中图分类号 S917.4文献标识码 A文章编号 0517-6611(2022)19-0102-04
doi:10.3969/j.issn.0517-6611.2022.19.024
开放科学(资源服务)标识码(OSID):
Construction and Analysis of COI Barcode of Twenty Fishes in National Aquatic Germplasm Resources Protection District of Luoma Lake
LI Da-ming LIU YangLIU Yan-shan1 et al
(1.Freshwater Fisheries Research Institute of Jiangsu Province,Jiangsu Key Laboratory of Inland Water Fishery Resources,Nanjing,Jiangsu 210017;2.College of Marine Science and Engineering,Nanjing Normal University,Nanjing,Jiangsu 210046)
Abstract The mitochondrial COI barcode of twenty fishes in national aquatic germplasm resources protection district of Luoma Lake were obtained by means of PCR amplification and sequencing.The results showed that the average content of bases in the COI gene sequence was A 24.4%,C 28.3%,T 28.5% and G 18.8%,respectively,and the A+T content (52.9%) was greater than the G+C content (47.1%).The intraspecific and interspecific genetic distances of the 20 fish species were 0-0.017 7 and 0.008 0-0.308 8,respectively,and the average genetic distances were 0.002 9 and 0.202 5,respectively.The interspecific genetic distance was 69.8 times of the intraspecific genetic distance.The average genetic distances within genus,family and order were 0.1500.206 1 and 0.236 4,respectively,indicating that the genetic distance values increased with taxonomic level.The results of molecular phylogenetic analysis showed that the 20 fish species were all monophyletic in the phylogenetic tree,which was consistent with the morphological classification results.This study is the first to construct COI barcodes of 20 species of fish in the Luoma Lake National Aquatic Germplasm Reserve,which can provide a scientific basis for the management of fishery resources and biodiversity protection in the reserve.
Key words DNA barcode;COI gene;Genetic distance;Species identification;Luoma Lake
我国是鱼类资源较为丰富的国家之一,近几十年来,受气候变化、环境污染、过度捕捞及水利工程的影响,我国渔业资源量锐减,物种多样性和遗传多样性下降,严重威胁渔业资源可持续发展利用[1-3]。快速、准确鉴定鱼类,是科学保护和管理鱼类资源的前提和基础。传统的鱼类鉴定是建立在鱼类形态特征基础之上,对分类人员的专业知识和鉴定能力要求较高。另外,由于鱼类的生物栖息环境多样,其形态结构易受到地理环境和不同发育阶段的影响,且鱼类“同种异形”和“异种同形”现象广泛存在,给鱼类种类鉴定造成困难[4]。
DNA条形码(DNA barcoding)技术是通过对一个标准目的基因的DNA序列进行分析而进行物种鉴定的技术,该技术具有不受样品性别、发育阶段、形态学变化限制的优点,且可以使物种鉴定过程实现标准化和信息化,近年来已成为生物分类学研究的热点[5-7]。线粒体COI基因的进化速度比核DNA快,所积累的遗传变异足以区分近缘种,且该基因容易被通用引物扩增,因此COI基因被选定为动物界物种鉴定的标准基因[8-10]。目前,COI基因作为DNA 条形码在鱼类的物种鉴定和多样性分析上也得到广泛应用,且已经建立了多个鱼类条形码数据库,为开展鱼类物种鉴定提供了便利[11-12]。
建立水产种质资源保护区是水产种质资源就地保护的一种有效形式,对保护水产种质资源发挥了重要作用[13]。经原农业部批准,骆马湖国家级水产种质资源保护区于2009年批准成立,保护区总面积3 160 hm 其中核心区面积1 000 hm 试验区面积2 160 hm2。保护区的主要保护对象是鲤鱼和鲫鱼[14]。近年来,有关骆马湖渔业资源调查的研究已有较多报道[15-17],但鲜见有关骆马湖鱼类DNA条形码的研究报道。该研究通过PCR扩增和测序,获得了骆马湖国家级水产种质资源保护区20种鱼类的COI条形码,并对COI基因序列特征、种内与种间遗传距离及分子系统进化关系进行分析,探索COI条形码在鱼类物种鉴定和分类中的适应性,以期为保护区鱼类资源保护和可持续开发利用提供基础资料。
1 材料与方法
1.1 试验材料
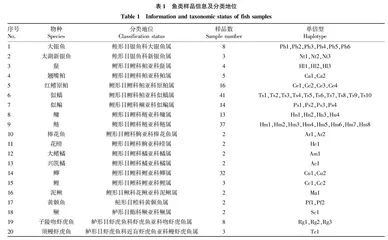
该研究鱼类样本来源于2019年4月和9月对保护区开展的渔业资源监测所得渔获物。根据《江苏鱼类志》对鱼类样本进行种类鉴定,所分析的20种鱼类样本信息及其分类地位见表1。剪取鱼类样品背部的肌肉组织置于95%乙醇中保存,用于后续基因组DNA提取。
1.2 DNA提取
采用TaKaRa公司的广谱型基因组DNA小量试剂盒提取鱼类样本的基因组DNA,并对DNA的完整性进行琼脂糖凝胶电泳检测,将DNA保存于-20 ℃冰箱备用。
1.3 PCR扩增和测序
采用鱼类DNA条形码通用引物进行PCR扩增,引物序列F1:TCAACCAACCACAAA GACATTGGCAC,R1:TAGACTTCTGGGTGGCCAAAGAATCA[18]。PCR反应体系为50 μL,其中包括0.4 μL Taq聚合酶(5 U/μL)、4 μL dNTP (2.5 mmol/L)、5 μL 10×PCR Buffer (含Mg2+),上、下游引物各2 μL (10 mmol/L),2 μL DNA模板,剩余部分超纯水补足。PCR反应程序:94 ℃预变性4 min;94 ℃变性40 s,54 ℃退火40 s,72 ℃延伸50 s,共30个循环;最后在72 ℃再延伸10 min。
PCR产物经1.5%琼脂糖凝胶电泳检测后,送上海生工生物工程股份有限公司纯化并进行双向测序,测序引物与扩增引物相同。
1.4 数据分析
利用BioEdit 7.0.5.3软件读取原始序列并进行人工校对,采用Clustal X1.83软件对序列进行多重比对分析,获得长度一致的同源序列,并在NCBI (http://www.ncbi.nlm.nih.gov/blast/ blast.cgi) 和BOLD (http://www.boldsystems.org)分别进行相似性检索。采用DnaSP 6.12.03软件定义序列的单倍型。利用MEGA 7.0软件统计序列的碱基组成、变异位点、简约信息位点、单一信息位点等,并基于Kimura′s 2-parameter双参数替代模型计算不同分类界元间的遗传距离。采用邻接法(neighbor-joining,NJ)构建单倍型分子系统进化树,并进行1 000次自展分支检验。
2 结果与分析
2.1 COI基因序列分析
通过PCR扩增、测序和分析,共获得20种鱼类的201条COI基因条形码序列,COI条形码长度为630 bp。COI序列碱基的平均含量分别为A 24.4%、C 28.3%、T 28.5% 和G 18.8%,碱基A+T的含量(52.9%)高于G+C的含量(47.1%),表现出明显碱基组成偏倚性,符合多数鱼类线粒体COI序列的碱基组成特征。另外,各密码子的碱基含量差异明显,第1密码子位点G+C的含量(58.8%)明显高于第2和第3密码子位点G+C的含量(分别为44.2%和38.5%)。所有COI序列没有插入或缺失位点,保守位点有379个,变异位点有251个,简约信息位点有239个,单一信息位点有12个。
2.2 各分类界元的遗传距离
基于MEGA 7.0 软件,采用K2P 模型计算各分类阶元的遗传距离(表2)。从表2可以看出,20种鱼类的种内遗传距离为0~0.017 7,平均值为0.002 9;多个鱼类的种内遗传距离均为0,比如花鱊骨、大鳍鱊、兴凯鱊、泥鳅、鳜和须鳗虾虎鱼,黄颡鱼的种内遗传距离最大。种间遗传距离为0.008 0~0.308 8,平均值为0.202 5,其中大鳍鱊和兴凯鱊的种间遗传距离最小,太湖新银鱼和泥鳅的种间遗传距离最大。种间遗传距离约为种内遗传距离的70倍,远大于Hebert提出的“10×规则”,即COI基因序列的种间遗传距离为平均种内遗传距离的10 倍以上才足以用来区分物种[19]。科内属间的遗传距离为0.016 9~0.213平均遗传距离为0.150其中鲤科的属和似鱎属间的遗传距离最小,鲤科的棒花鱼属和鱊属间的遗传距离最大。目内科间的遗传距离为0.202 5~0.209 7,平均遗传距离为0.206鲤形目的鲤科和鳅科间的遗传距离为0.202 5,鲈形目的虾虎鱼科和鮨科间的遗传距离为0.209 7。目间的遗传距离为0.192 6~0.264 7,平均遗传距离为0.236 4,其中鲤形目和鲑形目间的遗传距离最大,鲤形目和鲇形目间的遗传距离最小。