铜绿假单胞菌核糖体蛋白YggL的演化与功能分析
作者: 杨帆 沈威 王强
摘要 以公共数据库中15个原核模式生物的代表性菌株、假单胞菌属及其近缘属75个物种的代表性菌株以及708个铜绿假单胞菌的基因组为研究对象,分析了含有2个拷贝的核糖体蛋白YggL的基因,发现YggL可划分为2个分支(Clade),并探讨了其演化历程。在铜绿假单胞菌中,相比于Clade I YggL,Clade II YggL在抗生素处理下表达量显著降低,且与同属于核糖体大亚基组成蛋白的Clade II L31、Clade II L36共同表达。与Clade II YggL共表达的基因与膜蛋白紧密关联,编码核糖体蛋白的基因多拷贝现象可能通过影响核糖体状态,从而为膜蛋白折叠和转运提供更好的条件。
关键词 铜绿假单胞菌;基因演化分析;核糖体蛋白;膜蛋白
中图分类号 Q 78 文献标识码 A 文章编号 0517-6611(2025)04-0077-09
doi:10.3969/j.issn.0517-6611.2025.04.016
开放科学(资源服务)标识码(OSID):
Evolution and Function Analysis of Ribosomal Protein YggL in Pseudomonas aeruginosa
YANG Fan, SHEN Wei, WANG Qiang
(School of Life Sciences, Nanjing University, Nanjing, Jiangsu 210023)
Abstract This study systematically analyzed a double copied gene encoding the ribosomal protein YggL among representative strains of 15 model prokaryotic organisms, 75 representative strains of Pseudomonas and its closely related genues, as well as 708 genomes of Pseudomonas aeruginosa from public databases. YggL can be divided into 2 distinct branches (Clades). Their evolutionary history was explored. Compared to Clade I YggL, the expression of Clade II YggL significantly decreased under antibiotic treatment in Pseudomonas aeruginosa. Clade II YggL co expressed with Clade II L31 and Clade II L36, which are also components of the ribosomal large subunit. Genes co expressed with Clade II YggL were closely associated with membrane proteins, suggesting that the phenomenon of multiple copies of genes encoding ribosomal proteins may affect ribosomal status, thereby providing better conditions for membrane protein folding and transportation.
Key words Pseudomonas aeruginosa;Gene evolutionary analysis;Ribosomal protein;Membrane protein
作者简介 杨帆(1999—),女,河北张家口人,硕士研究生,研究方向:植物与微生物的基因演化。*通信作者,副教授,博士,硕士生导师,从事植物与微生物的基因演化研究。
收稿日期 2024-04-19
铜绿假单胞菌(Pseudomonas aeruginosa)是一种革兰氏阴性菌,广泛分布于土壤、动植物、临床等环境[1]。在临床环境中,铜绿假单胞菌已成为引发革兰氏阴性菌感染的重要原因,尤其在宿主免疫系统受损的患者中更为常见[2]。作为常见的条件致病菌之一,由铜绿假单胞菌造成的医院感染比例高达10.1%,可导致呼吸道、烧伤创面、泌尿道等严重感染,与许多疾病的高发病率和死亡率有关[3–5]。铜绿假单胞菌的基因组为5.5~7.0 Mbp,比大多数细菌都要大[1,6]。为了适应多元的生存环境,铜绿假单胞菌可通过祖先复制或者从其他物种水平基因转移的方式来获得更多与环境适应性相关的基因[7-8]。
生物的核糖体是由大、小两个亚基组成的复合结构,每个亚基包括核糖体RNA(rRNA)和多个核糖体蛋白(ribosomal proteins,RPs)[9]。原核生物的70 S核糖体由30 S小亚基和50 S大亚基组成,其中30 S亚基由1个16 S rRNA和约21种蛋白质组成,而50 S亚基由5 S、23 S rRNA和31种蛋白质组成[10]。在细菌中,核糖体蛋白不仅与核糖体形成和蛋白质翻译有关,还可以影响细胞生命活动的其他过程,如生长发育、细胞凋亡和衰老等[11]。同时作为许多重要抗生素的靶标,核糖体蛋白在医学领域的研究至关重要[12]。随着生物技术的不断进步和发展,人们对于核糖体蛋白的认识逐渐深入。有研究报道,一个新发现的核糖体蛋白YggL是大肠杆菌核糖体大亚基的组成部分,影响大小亚基组装成完整核糖体,拓展了对核糖体结构和功能的了解[13]。探究铜绿假单胞菌核糖体蛋白的进化来源和潜在的功能差异,有助于发掘其适应复杂环境及获得耐药性的分子机制。
笔者在来自厚壁菌门、放线菌门、衣原体门和变形菌门中15个模式生物的代表性菌株中鉴定了54个核糖体蛋白,发现铜绿假单胞菌的YggL、L31和L36各有2个拷贝,而其他细菌仅有1个拷贝。笔者聚焦于新发现的编码YggL的基因多拷贝现象,进行了系统的演化分析,旨在回答以下问题:①具有2个YggL拷贝是否是PAO1菌株所独有的,或者是铜绿假单胞菌或假单胞菌广泛拥有的?②2个YggL拷贝的进化起源是什么?③2个YggL拷贝是否分化出新的功能并参与不同的生物学过程?
1 材料与方法
1.1 蛋白鉴定
从NCBI数据库(https://www.ncbi.nlm.nih.gov/genome)下载γ-变形菌纲和模式菌株(reference genomes)的公开基因组信息。采用蛋白结构域鉴定方法,利用NCBIfam、PANTHER以及Pfam中的hmm文件作为种子文件,并使用HMMER软件[14]中的HMMsearch工具分析,设置参数为E-value=1e-5,进而完成对Ribosomal Protein Gene Database(RPG)数据库(http://ribosome.med.miyazaki u.ac.jp)列出的模式生物大肠杆菌的53个细菌核糖体蛋白及近年新发现的YggL的鉴定。
1.2 进化树的构建
通过HMMsearch鉴定出用于构建系统发育树的120个细菌蛋白(bac 120)[15]。使用MUSCLE软件[16]对bac 120和鉴定出的核糖体蛋白序列进行比对,利用IQ-TREE[17]构建系统发育树和蛋白进化树,并采用iTOL[18](interactive tree of life)进行标注和美化,以提高树形图的可视化效果。
1.3 基因组学分析
基因组学分析包括共线性分析和泛基因组分析。从NCBI核苷酸数据库(https://www.ncbi.nlm.nih.gov/nuccore)下载相应菌株的基因组组装文件及注释文件,用Clinker软件[19]进行共线性分析,以铜绿假单胞菌的模式菌株PAO1作为参照。使用PPanGGOLiN[20]对NCBI数据库公开的708株铜绿假单胞菌的基因组序列进行泛基因组分析,使用Gephi软件[21]对上述结果进行可视化。
1.4 蛋白结构预测
利用ColabFold[22]对YggL的2个拷贝进行了三维结构预测,在PyMOL[23]软件中进行2个拷贝之间的差异情况分析。
1.5 加权基因共表达网络分析(WGCNA)
从NCBI GEO数据库(https://www.pseudomonas.com)中下载铜绿假单胞菌PAO1在不同处理条件下的基因芯片数据。由于不同数据集在数据量和测序深度上存在差异,因此仅从23个GEO数据集中选取16个有Gene Expression Omnibus Series(GSE)原始数据进行后续分析。用R语言affy软件包[24]中的mas5函数对GSE原始数据进行标准化。根据处理条件将203个样本分成36个测试组,从这36个测试组中随机选择1、2或3个条件的组合,共得到7 806个不同的条件组合,去除未发现共表达情况的33个条件组合,总计条件组合为7 773个。用R语言WGCNA软件包[25]分别对3类核糖体蛋白的不同拷贝进行共表达网络分析,设定阈值>μ+2σ以筛选出具有实际统计学意义的共表达基因,以韦恩图的形式展示[26]。通过DAVID[27]进行GO分析,GO分析的结果用R中的ggplot2包[28]进行可视化。
2 结果与分析
2.1 核糖体蛋白YggL在细菌中的分布情况
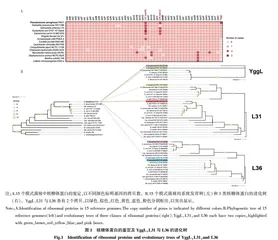
为全面了解核糖体蛋白在细菌中的存在情况,选取来自厚壁菌门、放线菌门、衣原体门和变形菌门中15个模式生物的代表性菌株,观察了RPG数据库收录的模式生物大肠杆菌的53个核糖体蛋白及近年来新发现的核糖体蛋白YggL在这些菌株中的分布模式。与其他代表性菌株只存在1个拷贝不同,在铜绿假单胞菌模式菌株PAO1中,核糖体蛋白L31、L36和YggL均鉴定到2个不同的拷贝(图1 A)。此前的研究已经明确,铜绿假单胞菌编码L31和L36的基因位点均存在2个拷贝的现象,并对二者的演化历程进行了探究,认为L31和L36第2个拷贝的来源可能由于水平基因转移事件导致,但难以排除变形菌进化基础上的复制[29]。值得注意的是,YggL基因也含有2个拷贝为该研究发现的现象。
通过构建15个模式菌株的系统发育树和3个核糖体蛋白的进化树,笔者发现YggL、L31与L36具有不同的分布模式。相比于同属于γ-变形菌纲的大肠杆菌(Escherichia coli)、肺炎克雷伯菌(Klebsiella pneumoniae)只含有1个YggL的情况,铜绿假单胞菌模式菌株PAO1基因组中含有2个YggL。与YggL不同,L31与L36分布十分广泛,不仅存在于γ-变形菌纲中,在厚壁菌门的金黄色葡萄球菌(Staphylococcus aureus)、放线菌门的结核分枝杆菌(Mycobacterium tuberculosis)、衣原体门的沙眼衣原体(Chlamydia trachomatis)等不同门类中也有所分布,且能够在大肠杆菌、铜绿假单胞菌、肠道沙门氏菌(Salmonella enterica)鉴定出多个不同的L31和L36(图1 B)。为便于描述,笔者将3类核糖体蛋白中存在于多数菌株的分支称为Clade I,只存在于少数菌株的核糖体蛋白分支称为Clade II。Clade I YggL由PA3046编码,Clade II YggL由PA1842编码;Clade I L31由PA5049/rpmE编码,Clade II L31由PA3601编码;Clade I L36由PA4242/rpmJ编码,Clade II L36由PA3600编码。