基于BSA-seq技术挖掘番茄耐裂果候选基因
作者: 冯汝龙 王彦刚 常晨晨 景涛 冯锡鸿 申太荣 董建力
摘要 [目的]明确番茄耐裂基因在染色体上的位置。[方法]以耐裂果番茄14FP5和易裂果番茄14FP26为试材,将二者杂交得到F1代,F1自交得到F2,分别构建2个极端性状的DNA混合池,采用BSA-seq方法进行定位。[结果]利用 BSA-seq 对F2 群体的2个极端混池进行基因组重测序分析,将番茄耐裂基因定位在5号染色体上,位于34 140 000~38 120 000 pb,区间大小为3.98 Mb,候选区间内共注释到38个基因,其中非同义突变基因4个。[结论]该研究结果可为番茄耐裂基因的精细定位提供数据支撑,为番茄耐裂分子育种奠定基础。
关键词 番茄;耐裂性;BSA-seq;基因定位;候选基因
中图分类号 S126 文献标识码 A 文章编号 0517-6611(2024)10-0120-05
doi:10.3969/j.issn.0517-6611.2024.10.025
Extraction of Tomato Cracking Tolerance Candidate Genes Based on BSA-seq Technique
FENG Ru-long,WANG Yan-gang,CHANG Chen-chen et al
(Ningxia Zhongqing Agricultural Science Co. Ltd., Yinchuan, Ningxia 750001)
Abstract [Objective] To determine the position of tomato crack tolerance gene on chromosome.[Method]Tomato 14FP5 which is resistant to cracking and tomato 14FP26 which is easy to crack are used as test materials,cross the two to get F1 generation, F1 self-cross to get F2,DNA mixing pools with two extreme traits were constructed respectively,BSA-seq method was used to locate the location.[Result]BSA-seq was used for genome resequencing analysis of 2 extreme mixed pools of F2 population.The tomato anti-cracking gene was initially located on chromosome 5,located between 34 140 000 and 38 120 000 pb,the interval size is 3.98 Mb,a total of 38 genes were annotated in the candidate region,including 4 non-synonymous mutant genes.[Conclusion]The results provide data support for the fine localization of tomato crack tolerance genes, and lay a foundation for tomato crack tolerance molecular breeding.
Key words Tomato;Cracking-resistant;BSA-seq;Assignment of genes;Candidate gene
基金项目 宁夏自然科学基金项目(2020AAC03508);宁夏回族自治区重大科技项目(2017DC55)。
作者简介 冯汝龙(1990—),男,山东济南人,助理农艺师,从事瓜菜育种与栽培研究。*通信作者,研究员,从事作物生物技术育种研究。
收稿日期 2023-07-02;修回日期 2023-07-20
番茄作为人们喜爱的果蔬,在我国有着“长寿果”的美誉。随着番茄种植面积的不断扩大,番茄在农业增效、农民增收及人们生活方面有着重要的位置。由于番茄在生产中易出现裂果现象,严重影响了番茄种植的经济效益。裂果已成为生产、销售中的一大危害。在生产中,通过栽培管理措施可以在一定程度上减轻番茄裂果的发生,却很难从根本上解决问题。因此,番茄耐裂品种的选育成为重要课题。
番茄裂果性属数量性状遗传,在开发特异性分子标记中存在成本高、周期长、效率低等缺点,较难获得特异性分子标记。群体分离分析法(bulked segregant analysis,BSA)通过表型差异构建分离群体快速筛选和定位目标性状相关基因[1]。BSA-seq 技术基于高通量测序技术,具有试验周期短、定位准确,开发的分子标记分布均匀、密度高等优点[2-3]。该方法在动植物功能基因定位研究中应用广泛[4-5]。
目前,关于高通量测序技术开发番茄耐裂特异分子标记鲜见报道。笔者在前期试验结果的基础上,利用BSA-seq技术对番茄耐裂基因进行定位,旨在为番茄耐裂分子育种奠定基础。
1 材料与方法
1.1 材料
供试易裂果番茄自交系14FP26和耐裂果番茄自交系14FP5,均来源于宁夏平罗中青种业公司。14FP26作母本与14FP5杂交,F1代自交产生F2分离群体,通过混合BSA法,分别选择20株耐裂单株和30株易裂单株构建混合DNA池的样品。
1.2 方法
1.2.1 裂果性鉴定。番茄裂果性鉴定采用苏彦宾等[6]的方法进行。
1.2.2
混池构建及测序。取F2极端耐裂和易裂各30株顶端幼嫩叶片1片,干冰保存寄往北京百迈克生物科技有限公司进行检测,采用改良的CTAB法提取DNA,将耐裂DNA和易裂DNA分别等量混合,构建2个后代极端DNA混池,利用 Illumina HiSeq 平台完成建库测序。
1.2.3 测序数据分析。
将构建的样品文库上机测序得到测序数据(raw reads),经Base Calling分析转化为原始测序序列(Sequenced Reads),其中含有带接头、低质量的Reads,为了保证信息分析质量,对Raw Reads进行过滤,得到Clean Reads,将Clean reads通过BWA 软件与番茄基因组进行比对。根据Clean read比对结果,利用 GATK 软件检测和过滤得到检测样品与参考基因组之间高质量的可信SNP位点和序列插入与缺失(InDel)位点。利用欧式距离法[7](euclidean distance,ED)和SNP-index 法计算与性状关联的候选区域。利用SNPindex 进行关联分析[7-8]。
1.2.4 候选区域筛选与功能注释。 关联分析结果合并取交集,用 BLAST[9]通过多个数据库(NR、Swiss-Prot、GO、KEGG、COG[10-11])深度注释候选区间内的编码基因,快速筛选候选基因。
2 结果与分析
2.1 番茄耐裂性鉴定
种植F2代群体单株共243株,从243株 F2群体中经表型鉴定获得裂果严重单株24株,中度裂果单株27株,轻度裂果单株55株,抗裂单株67株,中抗单株42 株,高抗单株28株。其抗性呈近似正态分布,这表明番茄耐裂性符合数量性状遗传。从裂果严重和高抗裂果中各取20株嫩叶,构建抗裂和易裂DNA混池。
2.2 测序数据量及数据质量
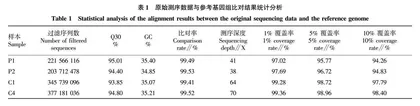
利用SLAF-seq技术对父本、母本和极端混池DNA进行测序,过滤后共获得了1 148 198 726条reads数据,其中,父本和母本分别为221 566 116、203 712 478条(表1),抗裂池和易裂池分别为345 739 096、377 181 036条。测序质量值 Q30为93.85%~95.01%,平均为94.52%,说明测序碱基错误率低,所获数据合格。测序获得GC含量在34.85%~35.40%,平均在35.13%。插入片段长度呈正态分布(图1),说明测序数据文库构建无异常。父本、母本和极端混池样品与参考基因组平均比对率为99.49%,基因组平均覆盖深度约为53.25X,1%覆盖率超过97.02%,5%覆盖率超过95.77%,10%覆盖率超过94.26%(表1),结果表明,测试数据符合标准,可进一步检测和筛选。
2.3 SNP和InDel变异检测与注释
SNP检测结果显示,共获得6 340 143个SNP,引起非同义突变的SNP共59 846个。其中,亲本P1、P2之间共获得2 710 004个SNP,非同义突变的SNP共24 737个,混合池C1、C4之间共获得3 630 139个SNP,引起非同义突变的SNP共17 767个。通过InDel检测,亲本之间共获得431 661个小片段插入与缺失(small InDel),混合池之间共获得 560 795个smal lnDel(图2)。大部分SNP和InDel分布在基因间、基因上游区段、基因下游区段。
2.4 关联分析
2.4.1 SNP关联分析。对SNP进行过滤后得到质量高的可信SNP位点1 487 748 个。采用ED算法和SNP-index算法对这些位点数据进行关联分析,得到4个染色体候选区段与番茄抗裂性相关联,区域所注释到的基因总长度为8.34 Mb。Chr05区段长度为3.98 Mb,位于34 140 000~38 120 000 bp,Chr05区段长度为3.98 Mb,位于40 640 000~44 620 000 bp,Chr011区段长度为0.08 Mb,位于3 740 000~3 820 000 bp(表2、图3)。根据SNP-index 算法关联阈值(0.95)判定,共得到2个与番茄抗裂性相关联的染色体区段,关联分布见图4,区段注释到的基因总长度为2.18 Mb,其中 Chr05区段长度最小,为0.06 Mb,位于42 240 000~42 300 000 bp。2种关联分析方法取交集,得到2个与性状相关的侯选交集区段,区段总长度为4.28 Mb(表2、图4)。
2.4.2 InDel关联分析。对 InDel位点进行过滤后得到质量高的可信InDel 位点192 616 个。根据ED关联阈值判定,共得到8个区段,总长度为12.54 Mb。采用与SNP关联分析时相同的方法(SNP-index),当置信度为0.99时,共得到5个区段,总长度为7.18 Mb。2种关联分析方法取交集,得到2个与番茄抗裂性相关的侯选区段,区段总长度为0.36 Mb(表3)。
2.5 候选区段筛选与基因注释
将SNP关联分析结果与InDel关联结果进行统计分析后取交集,InDel交集结果为空,该项目默认采用SNP的候选区间结果(表2),最终锁定番茄耐裂性候选区段在5号染色体上,位于34 140 000~38 120 000 pb,区间大小为3.98 Mb。利用 GO[12]、KEGG[11]等数据库对候选区段内的基因进行深度注释,有38个基因被注释,其中非同义突变基因4个。
利用番茄基因组数据库信息,利用 BlAST软件对候选区段内的基因进行NR、GO、KEGG、COG数据库进行深度注释,在候选区段内共注释到 38个基因,其中在 NR、trEMBL数据库中均注释到 38 个基因,Swiss Prot数据库中注释到29个基因,GO 数据库中注释到31个基因,KEGG数据库中注释到23个基因,COG数据库中注释到12个基因(表4)。