鹅掌楸基因组部分SNP标记在种源评价中的应用
作者: 陆叶 施季森
摘要 以6个鹅掌楸(Liriodendron chinense)种源和3个北美鹅掌楸(Liriodendron tulipifera)种源共计23个个体作为研究群体,选取11对真实SNP位点,对这253个SNP标记的PCR产物进行正反向Sanger测序。结果表明:在11个真实SNP位点中,8个SNP位点在中国鹅掌楸和北美鹅掌楸间呈现出多态性,其中3个位点是检测鹅掌楸和北美鹅掌楸种间分化的潜在标记。11个SNP标记在北美鹅掌楸种内都没有多态性。lm_ll_10205、lm_ll_10836、lm_ll_3853和lm_ll_8246 4个SNP位点在我国东部的湖南浏阳、浙江松阳和江西庐山3个种源间存在多态性,仅有lm_ll_10205位点在我国西部种源间存在多态性。前期开发的SNP标记可以用于鹅掌楸种源遗传多样性的研究。研究种源间SNP位点的变异特征为揭示鹅掌楸的起源、进化以及遗传变异多样性提供了科学数据。
关键词 北美鹅掌楸;鹅掌楸;SNP标记;SNP位点;种源
中图分类号 S792.21 文献标识码 A 文章编号 0517-6611(2024)21-0093-04
doi:10.3969/j.issn.0517-6611.2024.21.019
开放科学(资源服务)标识码(OSID):
Application of SNP Markers of Liriodendron Genome in Provenance Evaluation
LU Ye, SHI Ji-sen
(Key Laboratory of Forest Genetics & Biotechnology of Ministry of Education of China, Co-Innovation Center for Sustainable Forestry in Southern China, Nanjing Forestry University, Nanjing, Jiangsu 210037)
Abstract In this study, a total of 23 individuals were selected from 9 provenances with representative geographical distribution (including six Liriodendron chinense and three Liriodendron tulipifera provenances) as the study population. After PCR with 11 pairs of primers from real SNPs, Sanger sequencing was performed on the 253 SNP-labeled PCR products. The results showed that among the 11 real SNP loci, 8 SNP loci showed polymorphism between Chinese and North American provenances, and 3 of them were potential markers for detecting the interspecific differentiation of Chinese and North American provenances. None of the 11 SNP markers were polymorphic within the North American Liriodendron provenances. Four SNP loci (lm_ll_10205, lm_ll_10836, lm_ll_3853 and lm_ll_8246) showed polymorphism in Liuyang (Hunan), Songyang (Zhejiang) and Lushan (Jiangxi) species in eastern China, while only lm_ll_10205 showed polymorphism in western species of China. The results showed that the SNP markers developed in earlier studies could be used to study genetic diversity of Liriodendron provenance. The variation characteristics of SNP sites between provenances provide useful scientific data for revealing the origin, evolution and diversity of genetic variation of Liriodendron.
Key words Liriodendron tulipifera;Liriodendron chinense;SNP makers;SNPs loci;Provenance
基金项目 江苏省农业科技自主创新资金项目“耐盐型杂交鹅掌楸新品种培育及推广示范”(SCX(23)3628)。
作者简介 陆叶(1982— ),女,江苏镇江人,实验师,从事林木遗传育种研究。*通信作者,教授,博士生导师,从事林木遗传育种研究。
收稿日期 2023-12-29
木兰科(Magnoliaceae)鹅掌楸属(Liriodendron)现仅存2个种,即鹅掌楸[Liriodendron chinense(Hemsl.)Sarg.]和北美鹅掌楸(Liriodendron tulipifera Linn.),是典型的孑遗植物[1]。新生代第三纪,该属物种广泛分布于欧亚大陆和北美洲。第四纪冰川之后,鹅掌楸分布于我国长江流域和越南北部,分化为东部和西部两个支系,北美鹅掌楸自然分布于北美洲东南部,遗传上位于我国东西部鹅掌楸类群之间,更接近东部类群[2]。由于结实率低,加上人为对其适生环境的干扰,鹅掌楸在我国已被列为二类濒危保护物种[3]。鹅掌楸属物种不仅在林业产业方面具有重要的应用前景,而且也是林木遗传多样性、群体结构、系统进化及杂交育种等领域理论研究的理想材料[4-5]。
RAD-seq(Restriction-site associated DNA sequencing)技术是利用限制性内切酶对基因组DNA进行酶切,然后进行高通量测序[6],可作为一种有效的方法,进行高通量的SNP标记的开发[7]。目前已有很多研究基于RAD-seq技术对物种的遗传多样性和遗传结构进行分析。刘学锋等[8]对168份蓝花楹种质材料进行RAD-seq测序,获得45 552个高质量的SNP,分析表明,来自川渝地区的蓝楹花具有相对较近的亲缘关系。潘文婷等[9]用RAD-seq鉴定了北美和中国种源的鹅掌楸共4 454个SNP标记,对鹅掌楸的遗传多样性和遗传结构进行分析,认为鹅掌楸属遗传结构的形成与其地理隔离和片段化分布有关。
前期研究以北美鹅掌楸和鹅掌楸的杂交F1代和2个亲本为材料,利用RAD-seq技术已开发的大量可靠的SNP标记[10],笔者从中选取了11对真实SNP位点,以6个鹅掌楸种源和3个北美鹅掌楸种源共计23个个体为研究材料,分析SNP位点在不同种源间的变异特征,旨在为研究鹅掌楸物种分子水平上的变异提供科学依据。
1 材料与方法
1.1 试验材料
种源材料均来自国家林业和草原局世行贷款项目管理中心于1990年推广支持体系中的“阔叶树中心组”的科研项目建立的实验林。在鹅掌楸全分布区内抽样收集了15个地理种源的鹅掌楸种子,同时分别从美国密苏里(Missouri)、路易斯安那(Louisiana)、北卡罗来纳(North Carolina,北卡)、南卡罗来纳(South Carolina,南卡)、佐治亚(Georgia)引进该地区北美鹅掌楸种源的种子,翌年分别在四川邛崃、湖北京山、湖南桃源、江西分宜和福建邵武进行了地理种源研究试验。2014年4月从定植于湖北省京山县虎爪山林场的24年生的种源材料中选取了9个种源的材料,每个种源23株,共计23株作为后续试验材料。取样具体如下:湖南浏阳的3株鹅掌楸(1-10、1-13、1-23),浙江松阳的2株鹅掌楸(4-1、4-24),江西庐山的3株鹅掌楸(8-3、8-17、8-24),四川叙永的3株鹅掌楸(9-5、9-19、9-22),贵州松桃的3株鹅掌楸(11-12、11-15、11-22),云南勐腊的2株鹅掌楸(12-5、12-22),北卡罗来纳的3株北美鹅掌楸(15-19、15-21、15-23),南卡罗来纳的2株北美鹅掌楸(18-16、18-21),密苏里的2株北美鹅掌楸(19-16、19-21)。其中,来自湖南浏阳、浙江松阳和江西庐山的种源为我国东南部种源,来自四川叙永、贵州松桃和云南勐腊的种源为我国西南部种源,来自北卡罗来纳、南卡罗来纳和密苏里的种源为北美种源。
1.2 试验方法
1.2.1 基因组DNA的提取。
新鲜叶片用液氮冷冻,-80 ℃保存。采用天根试剂盒(DNAsecure Plant Kit,离心柱型)提取DNA,保存在-20 ℃冰箱中。
1.2.2 引物合成。
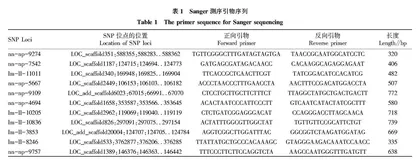
根据前期研究开发的鹅掌楸27个真实SNP位点[10],选取其中的11个SNP位点,利用鹅掌楸和北美鹅掌楸不同种源个体,对SNP位点在种群间的多态性进行研究,引物序列见表1。
1.2.3 PCR扩增体系。
用11对引物和KOD FX(Toyobo,KFX 101)DNA聚合酶对23个DNA样本进行PCR,将具有特异性条带的PCR产物进行Sanger测序。测序由上海英骏生物技术有限公司完成。用BioXM2.6软件比对测序结果。
PCR反应体系(50 μL)为:2×PCR Buffer for KOD FX 25 μL,2 mmol/L dNTPs 10 μL,10 μmol/L Primer 1.5 μL,KOD FX DNA polymerase 1 μL,DNA 5 μL,ddH2O 6 μL。PCR 反应程序如下:94 ℃预变性 2 min;98 ℃变性10 s,Tm 5 ℃退火 30 s,68 ℃延伸 1 min,36 个循环;最后4 ℃保温。
2 结果与分析
2.1 DNA质量检测
对提取的DNA进行琼脂糖电泳检测,如图1所示,DNA条带清晰完整,无明显弥散条带,表明DNA质量好且纯度较高。用Nanodrop检测DNA样品的OD260/OD280值,均在1.8~2.0。高质量的DNA达到后续试验要求。
2.2 PCR产物检测
PCR产物经过琼脂糖凝胶电泳检测,将符合预期的目的条带切胶回收、纯化后进行Sanger测序验证。图2为部分引物在不同个体间的PCR扩增结果。
2.3 SNP位点检测