日本沼虾体重和出肉率联合GWAS分析
作者: 刘帅 蒋丽
摘要 [目的]定位筛选影响日本沼虾体重及出肉率性状的候选基因。[方法]收集成熟期且表现健康的日本沼虾,记录体重、出肉率,提取DNA进行测序并进行基因分型,获得单核苷酸多态性(Single-nucleotide polymorphisms,SNP)标记数据,利用多性状联合的全基因关联分析(Genome-wide association study,GWAS)方法检验与体重、出肉率性状存在显著相关的SNP位点,根据SNP的物理信息实现候选功能基因的筛选和定位。[结果]联合分析共定位到6个SNP位点,检测效率远高于单一分析的1个SNP,根据6个显著位点共定位到5个候选功能基因, 除1个功能未知的新基因外,剩余4个基因都与机体代谢、发育相关。[结论]完成了日本沼虾体重、出肉率性状相关基因的定位,可为日本沼虾生长代谢的基因组水平揭示提供理论基础,也可为日本沼虾的其他数量性状研究提供思路。
关键词 日本沼虾;全基因组关联分析;体重;出肉率
中图分类号 S 917.4 文献标识码 A
文章编号 0517-6611(2023)02-0108-03
doi:10.3969/j.issn.0517-6611.2023.02.027
Combined GWAS Analysis of Body Weight and Meat Rate in Macrobrachium nipponense
LIU Shuai1.2,JIANG Li2
(1.National Demonstration Center for Experimental Fisheries Science Education,Shanghai Ocean University,Shanghai 201306;2.Key Laboratory of Aquatic Genomics,Ministry of Agriculture and Rural Affairs,Chinese Academy of Fishery Sciences,Beijing 100141)
Abstract [Objective] In order to screen candidate genes that affect the traits of body weight and meat yield in Macrobrachium nipponense.[Method]Collecting the Macrobrachium nipponense performed mature and healthy,recording the body weight and meat rate,extracting the DNA,product database of single-nucleotide polymorphisms (SNP) maker through sequencing and genotype for the extracted DNA.Employing the method of multi-trait combined whole gene association study (GWAS) to find out the significant SNPs related to the trait of body weight and meat ratio,and finished screening and location of candidate functional genes according to the physical information of detected SNPs.[Result] The combined analysis for body weight and mean rate was located 6 SNPs,its detection efficiency was much higher than that total of 1 SNP in the single analysis for each trait.And a total of 5 candidate functional genes were located,most of them were related to body metabolism and development except one new function gene with the power of unknown according to the 6 significant SNPs.[Conclusion]This study completed the mapping of genes related to weight and meat rate traits in Macrobrachium nipponense,which can provide theoretical support for revealing growth and metabolism of Macrobrachium nipponense at the genomic level,and provide ideas for the study of other quantitative traits of Macrobrachium nipponense.
Key words Macrobrachium nipponense;GWAS;Body weight;Meat rate
基金项目 国家重点研发计划项目(2018YFD0901303)。
作者简介 刘帅(1996—),男,河南鹤壁人,硕士研究生,研究方向:水产动物遗传育种。通信作者,副研究员,博士,从事水产动物遗传育种研究。
收稿日期 2022-02-08;修回日期 2022-03-30
日本沼虾(Macrobrachium nipponense)俗称青虾、河虾,属于节肢动物门(Arthropoda)甲壳纲(Crustacea)十足目(Decapoda)长臂虾科(Palaemonidae)沼虾属(Macrobrachium),广泛分布于我国及东南亚大部分地区,由于其肉质鲜美,营养丰富,是我国重要的淡水养殖虾种之一。雌雄差异及群体均匀度低是制约集约化养殖产量的主要因素,因而,从基因组水平对其体重相关的基因进行解析,进而培育具有较高均匀度且增重快的新品种对其养殖业发展具有战略意义。
全基因组关联分析(genome-wide association study,GWAS)是现代分子育种的重要辅助手段,通过单核苷酸多态性(single nucleotide polymorphism,SNP)与目标性状之间的关联检验,实现相关基因的定位,在水产动物育种中发挥着重要作用。经典的GWAS只针对单一性状进行分析,但当不同性状间存在遗传相关时,考虑性状间协方差的多性状分析相较于单性状分析具有更高的检验功效[1]和参数估计的精度[2]。已有研究证明,多性状联合 GWAS分析对显著遗传变异的检测能力更强[3],且更具生物学意义[4]。基于此,笔者对日本沼虾体重和出肉率性状进行联合分析,实现对相关基因的定位和筛选,以期为日本沼虾生长增重的基因表达解析提供理论支撑,进而促进其养殖业发展。
1 材料与方法
1.1 试验群体及表型测量 试验用虾养殖于中国水产科学研究院淡水渔业研究中心宜兴大浦基地,收集日龄52 d的成熟虾,随机挑选200尾健康虾(雌雄各100尾)编号,记录体重,收集虾尾部的肌肉并称重,尾部肌肉与体重的比值为出肉率。此外,收集的肌肉需转入冻存管中于-80 ℃保存,以便进行DNA提取。
1.2 DNA提取、测序及基因型数据的获取
以收集的肌肉组织为材料,根据酚-氯仿法[5]提取DNA,使用1%琼脂糖凝胶评估提取DNA质量,并将其浓度调整至2.5 μg/μL,最终在北京华大基因完成序列测定。原始测序数据经工具包NGS QC Toolkit[6]质控过滤后,通过软件BWA[7]与日本沼虾参考基因组(GCA_015104395.1)进行对比分析,通过软件Picard(https://github.com/broadinstitute/picard)去除对比结果中的重复序列,最终由软件GATK(https://github.com/broadinstitute/gatk)实现SNP位点的筛选,从而完成基因分型。获得的分型数据需要通过PLINK[8]软件进一步质控和过滤,去除样本分型成功率≤90%,最小等位基因频率≤5%的位点并生成二进制基因型数据。
1.3 全基因组关联分析
分别对体重、出肉率性状进行单性状的全基因组关联分析,并对2个性状进行多性状联合分析,其中单性状分析使用GEMMA[9]软件中的单性状混合模型分析,多性状联合分析采用GEMMA软件中的多性状线性混合模型。以Bonferroni校正阈值[10]为界限筛选与性状显著相关的SNP位点,并通过R语言绘制曼哈顿和Q-Q图。
1.4 基因注释
根据检验显著SNP位点于参考基因组上的物理位置,提取其上下游区域250 kb范围内的序列,通过软件KOBAS(http://kobas.cbi.pku.edu.cn/kobas3)调整参数“-e 1e-5”实现对提取序列的注释,根据NCBI数据库及文献检索实现注释的功能注释。
2 结果与分析
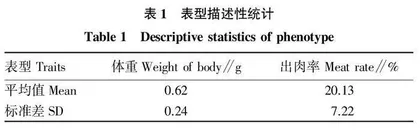
2.1 表型及基因型描述统计
剔除体重、出肉率表型数据中的缺失项后共获得181尾虾的表型数据(表1),性状间的相关关系及分布见图1~3。基于181尾虾分型获得的基因型数据中剔除分型成功率≤90%及最小等位基因频率≤5%的位点后,共获得7 793个高质量SNP位点。
2.2 全基因组关联分析
对体重、出肉率进行单一性状的全基因组关联分析及两性状的联合分析,结果如图4所示。观察图4 d~图4f,根据统计检验概率P值拟合的实际曲线均与理论
曲线有较高的一致性,且基因组控制值(λ)位于1.00附近[11],说明关联检验的统计准确性较高,进而可说明曼哈顿图中检测到的SNP具有较高的可信度(图4a~c),水平线表示Bonferroni校正阈值(0.05/7793,6.42e-06)分布于水平线上方的位点具有与性状的显著相关性。由图4a可知,在体重单性状关联分析中,仅检测到1个SNP位点与之相关;在出肉率的单性状关联分析中(图4b),并未检查测到显著SNP位点;在两性状的联合分析中(图4c),共检测到6个SNP位点,且体重单性状分析检测到的SNP位点也被包含在内,这证明多性状联合分析对SNP检测的效率具有显著提升作用。
2.3 候选基因
根据日本沼虾体重和出肉率联合分析获得的显著SNP 位点为探针,提取其所在日本沼虾全基因组位置上下游250 bp区域内的序列,通过数据库kobas完成候选基因的在线注释与鉴定,共鉴定出5个候选基因(表 2),分别为Jrkl(JRK like)、Tep2(Thioester-containing protein 2)、ZNF84(zinc finger protein 84)、CG12299和swm(second mitotic wave missing)。通过KEGG通路注释分析,这些候选基因主要参与基因表达调控、转录调控、细胞信号转导、蛋白酶解、内肽酶分析及先天性免疫系统发育等生物学过程,在机体分化发育方面发挥着重要作用。
3 讨论
随着测序成本的下降及多种水产动物基因组全测序的完成,GWAS技术逐渐应用于水产动物的基因定位及育种领域。吴碧银等[12]通过GWAS分析技术发现了影响低氧环境下鲤耐受性的23个候选基因。陈小明等[13]利用GWAS检测到26个与大黄鱼耐高温相关的基因。徐鸿飞等[14]对红罗非鱼低温体色变异现象进行GWAS分析,发现了19个与之相关的功能基因。