不同悬钩子属植物ITS序列的多态性分析
作者: 高柱 郭芙蓉 杨静远 汪琪
摘要 以13份悬钩子属植物DNA为模板,用PCR技术克隆获得其ITS序列。测序结果分析表明,ITS序列总长在462~464 bp,其中5.8S、ITS1和ITS2序列长度分别为164、208~210、254~255 bp。序列差异比对分析表明,5.8S序列相对保守,不同悬钩子属植物仅有1个碱基差异;ITS2序列变异较大,GC含量在53.73%~55.12%,存在32个变异位点,其中单核苷酸差异位点14个。基于ITS2序列的系统进化树分析表明,13份悬钩子种质资源可分为4簇,其中簇Ⅰ包含掌叶覆盆子种质资源A9、A29和甜叶覆盆子种质资源GH14,簇Ⅱ为HH2、W5和JF9等山莓种质资源,簇Ⅲ为红树莓及黑莓种质资源,簇Ⅳ为TW1、GA1、S和GB3等插田泡种质资源。ITS2序列聚类分析的结果与形态特征分类的结果是一致的,表明外缘标准的ITS2序列对悬钩子属植物资源进行物种分类和鉴定是可靠的。
关键词 悬钩子属;ITS序列;克隆;分子鉴定
中图分类号 S663.2 文献标识码 A 文章编号 0517-6611(2023)19-0095-05
doi:10.3969/j.issn.0517-6611.2023.19.020
Polymorphism Analysis of ITS Sequences in Different Rubus Genera
GAO Zhu1, GUO Fu-rong2, YANG Jing-yuan2 et al
(1. Anhui Guoke Testing Technology Co., Ltd., Hefei, Anhui 230041; 2. School of Life Sciences, Anhui Agricultural University, Hefei, Anhui 230036)
Abstract Thirteen germplasm resources in the genus Rubus were used as materials for internal transcribed spacer (ITS) sequence cloning by PCR methods. Sequence alignment analysis revealed that the full length of ITS sequences ranged from 462 to 464 bp. The sequence lengths of 5.8S, ITS1 and ITS2 were 164 bp, 208-210 bp and 254-255 bp, respectively. The comparative analysis of sequence differences showed that the 5.8S sequence was relatively conservative, with only one base difference among different rubus genera. ITS2 sequence was highly variable, with GC content ranging from 53.73% to 55.12%, and there were 32 variation sites, including 14 single nucleotide difference sites. Phylogenetic tree analysis based on ITS2 sequence showed that the germplasm resources collected can be divided into four sub-clusters. Among these germplasm resources, the genetic distance between Rubus chingii Hu A9, A29 and Rubus suavissimus S. Lee GH14 were divided into sub-cluster Ⅰ. Rubus corchonfolius L. F. HH2, W5 and JF9 were divided into sub-cluster Ⅱ, and red raspberries and black berries were divided into sub-cluster Ⅲ. And Rubus coreanus Miq. TW1, GA1, S and GB3 were divided into sub-cluster Ⅳ. The ITS2 analysis is consistent with the results of morphological feature classification, indicating that the ITS2 sequence is reliable in the classification and identification of species in the genus Rubus.
Key words Rubus;ITS sequence;Cloning;Molecular identification
悬钩子属(Rubus)为蔷薇科(Rosaceae)蔷薇亚科(Rosoideae)中的一个属,落叶稀常绿灌木、半灌木或匍匐草本,多年生宿根植物。据统计,该属植物目前已发现700余种,分布于全世界,主要产地在北半球温带,少数分布到热带和南半球,我国有204种104变种[1]。悬钩子植物在我国西南地区,特别是四川、广东、广西、福建及云南等[2]地区有较多分布,占目前全国总悬钩子属植物种数的60%以上。多数悬钩子属植物的果实、种子、根及叶可入药,具有驱寒祛湿、止血止痛、清热解毒、活血化瘀及固肾涩精等功效[3];有些种类的果实多浆,味甜酸,可供食用[4]。
悬钩子属植物种类繁多[5],植物分类学家对其分类进行了大量研究工作,但不同学者在悬钩子属植物的范围划分和亚属的分类方面有不同见解。1979年刘金[6]将悬钩子属植物分为实心莓亚属、刺毛莓亚属、空心莓亚属、大花莓亚属和软枝莓亚属5个亚属。俞德浚等[7-9]以悬钩子属植物主要器官性状特征及细胞学资料为依据,将已发现的194种悬钩子属植物分为8组24亚组,其中,空心莓组(Sect.Idaeobatus)和木莓组(Sect.Malachobatu)植物包含种类较多。空心莓组(Sect.Idaeobatus)植物又分为11个亚组,共83种50个变种;木莓组(Sect.Malachobatu)植物又分为13个亚组,85种35个变种。亚组之间的分类主要依据该植物的叶型、花序及叶片性状等生物学特征[10]。近年来,我国植物学工作者在各国家和地区又陆续发现许多新种,如腺果悬钩子(R.glandulosocarpus)、武夷悬钩子(R.jiangxiensis)、铅山悬钩子(R.yanshanensis)、少花悬钩子(R.spananthus)和九仙莓(R.yanyunii),补充并完善了我国悬钩子属植物的分类系统[11-14]。
悬钩子属植物因其种类繁多,变异性大,类型复杂,而且存在无融合生殖现象,并伴有多倍体出现,仅依据外部形态特征进行分类比较困难。笔者利用DNA条形码技术,探索悬钩子属植物ITS序列的特征,揭示其系统进化关系,为悬钩子属植物的分类鉴定提供科学依据。
1 材料与方法
1.1 材料
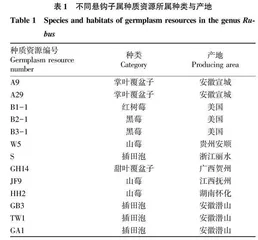
用于ITS分析的13份悬钩子属核心种质资源分别采集自安徽、贵州、浙江、广西、江西和湖南,定植于安徽农业大学国家高新技术农业园,种质信息见表1。
1.2 悬钩子属植物DNA的提取
研钵、枪头、离心管等器材均经高温灭菌处理,利用植物DNA提取试剂盒(艾德莱生物公司,北京)提取悬钩子属植物的基因组DNA,具体操作参照试剂盒说明书。检测合成DNA样品于-20 ℃保存备用。
1.3 悬钩子属植物ITS序列的克隆
克隆ITS序列所用的PCR引物分别为ITS-F:GGAAGGAGAAGTCGTAACAAGG、ITS-R:CTTTTCCTCCGCTTATTGATATG,引物委托公司合成。PCR反应体系参照2×Taq Master Mix说明书(擎科生物科技有限公司,南京)。反应程序为98 ℃预变性3 min,变性10 s;55 ℃复性10 s;72 ℃延伸10 s,35个循环;72 ℃延伸2 min。胶回收采用MiniBEST Agarose Gel DNA Extraction Kit试剂盒(宝生物工程有限公司,大连),胶回收产物委托公司测序。
1.4 悬钩子属植物ITS序列的生物信息学分析
采用基于隐马尔科夫模型的注释方法(http://its2.bioapps.biozentrum.uni-wuerzburg.de/),去除测序序列的5.8S和ITS1序列,获得ITS2间隔区序列。利用NCBI网站BLAST工具,对ITS2序列进行比对检验。
利用DNAMAN软件对5.8S、ITS1和ITS2序列进行比对;利用BioEdit 7.0软件对ITS2序列单个碱基进行分析;利用MEGA7软件对ITS2序列进行分析,利用邻接法(Neighbor Jioning,NJ)构建系统发育树。构建ITS2系统进化树的近缘植物的相关序列见表2。
2 结果与分析
2.1 悬钩子属植物基因组DNA的ITS序列克隆及鉴定
选取不同悬钩子属种质资源13份,开展基因组DNA的ITS克隆和分析。以不同供试材料DNA为模板,进行了ITS序列的PCR扩增,将克隆的产物进行琼脂糖凝胶回收检测,结果见图1。由图1可知,核心种质资源均获得单一的PCR条带,条带大小约500 bp。
对供试材料PCR产物进行纯化回收,对测序结果进行5.8S、ITS1和ITS2序列分析(表3)。结果显示,13份悬钩子属种质资源ITS区域的长度在462~464 bp。ITS1的序列长度在208~210 bp,GC含量为55.29%~58.17%;ITS2的序列长度在254~255 bp,GC含量为53.73%~55.12%;13份悬钩子属种质资源的5.8S序列相对保守,长度均为164 bp,GC含量为54.27%或54.88%。
2.2 悬钩子属植物5.8S序列
用DNAMAN软件对ITS序列的5.8S区域进行分析,结果表明,悬钩子属植物的5.8S序列是高度保守的一个变异位点(124位的碱基A变成G)。其中,A9、A29、W5、GH14、JF9、HH2的5.8S区域序列一致为TAAACGACTCTCGGCAACGGATATCTCGGCTCTCGCATCGA-TGAAGAACGTAGCGAAATGCGATACTTGGTGTGAATTGCAG-AATCCCGTGAACCATCGAGTCTTTGAACGCAAGTTGCGCCC-AAAGCCATTAGGCCGAGGGCACGCCTGCCTGGGCGTCACAC;B1-1、B2-1、B3-1、S、GB3、TW1、GA1的序列一致为TAAACGACTCTCGGCAACGGATATCTCGGCTCTCGCATCGATGAAG-AACGTAGCGAAATGCGATACTTGGTGTGAATTGCAGAATCC-CGTGAACCATCGAGTCTTTGAACGCAAGTTGCGCCCGAAG-CCATTAGGCCGAGGGCACGCCTGCCTGGGCGTCACAC。
2.3 悬钩子属植物ITS2序列
利用BioEdit 7.0和DNAMAN软件对悬钩子属植物的ITS2序列进行比对和差异分析(图2)。结果显示,13份悬钩子属种质资源的ITS2序列变异较大,存在碱基缺失的现象。其中JF9、GB3和TW1在+14 bp处存在1个碱基缺失,B1-1、B2-1和B3-1在+86 bp处存在1个碱基缺失。另外,除了A29、B1-1、S和HH2之外,其余种质资源在+123 bp处均存在1个碱基缺失。ITS2含有223个保守位点,32个信息位点,其中变异位点18个,单个核苷酸差异位点14个。