基于线粒体COⅠ基因滁州地区克氏原螯虾养殖群体遗传多样性分析
作者: 余红喜 刘帆 朱国美 任信林 刘鑫鑫 凌武海 苏时萍
摘要 [目的]了解安徽滁州市克氏原螯虾养殖群体的遗传学特征。[方法]基于线粒体细胞色素氧化酶亚基I(COⅠ)基因序列,采用直接测序法分析8个克氏原螯虾养殖群体的遗传多样性变化、群体结构和遗传分化情况。[结果]8个养殖群体共有 25个变异位点,包括15个简约信息位点和10个单碱基变异位点;序列中(A+T)的含量(67.0%) 明显高于(G + C) 的含量( 33.0% ),具有明显的(A+T)碱基偏倚。遗传多样性分析显示,全部样本的单倍型多样性指数为0.373 14,核苷酸多样性指数为 0.001 72,呈现低单倍型多样性和低核苷酸多样性,且单倍型之间不具有明显的地理分化特征,不同养殖群体之间混杂在一起。群体间遗传分化系数FST为-0.023 56~0.122 14,分化程度极低。[结论]滁州市8个克氏原螯虾养殖群体的遗传多样性处于较低水平,建议扩大亲本选种范围,避免近亲繁殖和盲目交换亲本。
关键词 克氏原螯虾;线粒体DNA;COⅠ基因;遗传多样性
中图分类号 S 966.12 文献标识码 A 文章编号 0517-6611(2022)21-0111-03
doi:10.3969/j.issn.0517-6611.2022.21.026
开放科学(资源服务)标识码(OSID):
Analysis of Genetic Diversity of P.clarkii Culture Population in Chuzhou Based on Mitochondrial COⅠ Gene
YU Hong-xi LIU Fan2, ZHU Guo-mei1 et al
(1.Chuzhou Agricultural and Rural Technology Promotion Center, Chuzhou, Anhui 239000;2.College of Animal Science and Technology,Anhui Agricultural University,Hefei,Anhui 230036)
Abstract [Objective]To research the genetic characteristics of farmed Procambarus clarkii population in Chuzhou City,Anhui Province. [Method]We compared nucleotide sequences of mitochondrial cytochrome oxidase subunit I (COⅠ) gene of mitochondrial DNA (mtDNA) from eight farmed populations to investigate changes in genetic diversity,population structure and genetic differentiation. [Result]The results showed that 8 populations had 25 mutation sites,including 15 parsimony information sites and 10 single-base mutation sites;the content of (A + T) (67.0%) was significantly higher than (G + C) (33.0%),with obvious (A+T) base bias. Genetic diversity analysis showed that the haplotype diversity index of all samples was 0.373 14,and the nucleotide diversity index was 0.001 72. The 8 populations showed low haplotype diversity and oligonucleotide diversity,moreover,the haplotypes do not have obvious geographic differentiation characteristics,but are mixed together among those farmed groups. The coefficient of genetic differentiation among populations FST was -0.023 56-0.122 14,and the degree of differentiation was extremely low. [Conclusion]The genetic diversity of 8 populations of farmed Procambarus clarkii population in Chuzhou City is at a relatively low level. It is suggested to expand the selection range of parents and avoid inbreeding and blind exchange of parents.
Key words Procambarus clarkii;Mitochondrial DNA (mtDNA);CO I gene;Genetic diversity
克氏原螯虾(Procambarus clarkii),隶属甲壳纲十足目螯虾科原螯虾属,俗称小龙虾。自20世纪30年代传入我国,在长江流域扩繁[1],其味道鲜美,高蛋白低脂肪[2],生命力顽强,适应性广,繁殖能力强,对水质要求不高,现已遍布于我国中东部地区十余省[3],成为我国稻田养殖的主要品种,养殖面积总量大,产量居淡水虾类之首[4]。由于近几年克氏原螯虾养殖面积和规模不断扩大,受其逆向选择及种苗自繁自育的养殖模式等人为因素的影响,使得克氏原螯虾个头逐年变小,病害频发,种质退化严重,群体遗传多样性受到严峻考验[5]。为了恢复与保护克氏原螯虾种质资源,提供优良品种的选育科学依据,并提高经济效益,研究克氏原螯虾遗传多样性与遗传结构至关重要。
线粒体DNA(mitochondrial DNA,mtDNA)是保守的共价闭合的双链DNA分子,具有结构简单、进化速度快、无重组、多拷贝、母系遗传等特点。线粒体细胞色素氧化酶亚基I(COⅠ)作为蛋白编码基因的一员,在分子标记上是研究最为清楚的线粒体基因之一,也有学者认为,相比其他线粒体基因,COⅠ基因是更为理想和常用的分子标记[6]。徐浩文等[7]分析了8个地理种群红条毛肤石鳖COⅠ基因的遗传多样性,单倍型遗传多样性数据显示,多样性数值较高,核苷酸多样性较低,结果表明,红条毛肤石鳖北方群体的遗传多样性与南方群体存在差异,2个群体应该分别采取相应措施来保护遗传多样性;李大命等[8]基于线粒体COⅠ基因序列分析江苏省4个太湖新银鱼群体遗传多样性,结果表明4个太湖新银鱼种群偏离中性进化,历史上经历过种群扩张;刘士力等[9]分析了红螯螯虾养殖群体线粒体 COⅠ基因序列的遗传结构,结果表明,5个红螯螯虾养殖群体的遗传多样性存在差异,群体间存在着一定的基因交流;轩中亚等[10]基于线粒体COⅠ序列分析中华绒螯蟹养殖群体与野生群体遗传多样性与遗传结构,发现野生中华绒螯蟹遗传多样性较低,尤其是在长江水系,不同地理群体之间的种质混杂情况较为严重,反映了过度捕捞对种质资源的极大破坏。然而,有关克氏原螯虾养殖群体线粒体COⅠ基因序列遗传多样性的研究鲜见报道。
安徽省滁州市稻田养殖克氏原螯虾至今已有25年,养殖群体分布广泛,也是最早开展克氏原螯虾苗种繁育的地区之一,以该地区为代表,研究克氏原螯虾养殖群体遗传多样性具有良好的代表性。笔者基于线粒体COⅠ基因序列,分析了安徽滁州市8个克氏原螯虾养殖群体的遗传多样性与群体结构,探究其养殖群体的遗传多样性现状,为更高效地开发利用安徽省克氏原螯虾种质资源及开展种质资源评估提供参考依据和科学指导。
1 材料与方法
1.1 试验材料
样品采自安徽省滁州市,分别为全椒县潘氏生态农业发展有限公司(F1)、全椒县赤镇龙虾经济合作社(F2)、全椒县金顺家庭农场(F3)、全椒县银花家庭农场(F4)、定远县曹永明家庭农场(F5)、定远锦鸿种养殖专业合作社(F6)、定远县成海家庭农场定远成海生态农场(F7)、定远县许令国生态种养殖家庭农场(F8),共8个群体200个样本。取其背部肌肉组织置于95%乙醇中,4 ℃保存,用于线粒体DNA抽提。
1.2 试验方法
1.2.1 线粒体DNA的抽提。肌肉组织使用YEASEN动物组织直接PCR试剂盒抽提DNA,-20 ℃保存。
1.2.2 COⅠ序列的扩增、测序。COⅠ引物序列[11]:LCO1490:5′-GGTCAA CAAATCATAAAGATATTGG -3′;HC02198:5′- TAAACTTCAGGGTGACCAAAAAATCA -3′,委托上海桑尼生物科技有限公司进行引物合成。 PCR反应体系为40 μL,包括2×Tissue Direct PCR Mix 20 μL,20 mol/L上下游引物各1 μL,DNA模板2 μL,ddH2O补足至 40 μL。扩增程序:94 ℃预变性5 min;94 ℃变性30 s,54 ℃退火30 s,72 ℃延伸40 s,共30个循环;最后一个循环结束后,72 ℃延伸10 min。
PCR扩增产物经凝胶电泳初步确定片段长度后,由上海桑尼生物科技有限公司进行产物纯化、测序。
1.2.3 遗传多样性和群体分化分析。
序列的比对、碱基组成、遗传距离使用MEGAX[12]进行分析。序列的变异位点、核苷酸多样性(Pi)、单倍型多样性(Hd)、分化系数(FST)使用DNAsp 5.0[13]进行分析。
1.2.4 群体结构分析。
单倍型网络图使用POPART[14]构建。邻接法系统进化树使用MEGAX[12]构建,并进行1 000次的Bootstrap检验。
2 结果与分析
2.1 遗传多样性和群体分化分析
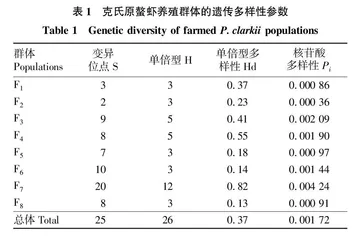
COⅠ测序数据经人工校正、比对、去除两端冗余序列和BLAST验证序列同源性后,共获得182条长度为655 bp的COⅠ序列用于下游分析,182条序列共检出25个变异位点,包括15个简约信息位点和10个单碱基变异位点;碱基平均含量为T(40.0%),C(13.0%),A(27.0%)和G(20.0%),具有明显的(A+T)碱基偏倚。遗传多样性分析显示(表1),多数群体具有较低的单倍型多样性(0.13 ~0.41 < 0.50),仅F7组拥有较高的单倍型多样性(Hd=0.82)。核苷酸多样性与单倍型多样性趋势一致,但所有群体均较低。总体来说,8个群体整体呈现低单倍型多样性及低核苷酸多样性的遗传多样性现状。
群体分化分析显示(表2),8个养殖群体间发生了不同程度的分化,从轻微分化(FST<0.05)到中等分化(0.05<FST<0.15)不等。其中,高分化系数主要集中在F7组与其他组之间,如F7组与F1组分化系数最高(FST=0.122 14),而其他组之间分化系数较低,多数低于0.05,可以认为没有分化,如F6组与F8组分化系数最低(FST =-0.023 56)。此外,相同的趋势在群体间遗传距离中也得到了验证。
2.2 群体结构分析
邻接法系统发育树分析显示(图1),个体并未按照养殖群体地理位置分布聚集成相应的分支,反而混杂在一起,且个体间遗传距离较近。单倍型网络图显示(图2),共检出26种单倍型,其中,Hap_1、Hap_2、Hap_4、Hap_7、Hap_15为多群体共享单倍型;Hap_2在群体共享单倍型中占主要分布,其余均为群体内独有单倍型;F7组群体拥有最多的单倍型(12个)。单倍型的分布方式与系统发育树趋势相同,并未依据地理位置而聚集,而是通过Hap_2替换数个碱基后相连接,多数单倍型为群体内部独有。