不同连作年限西红花种植土壤微生物多样性及网络特征的差异
作者: 李孟芝 封貌 高丽 梁鹏 谢锐 申皓天 黄显章
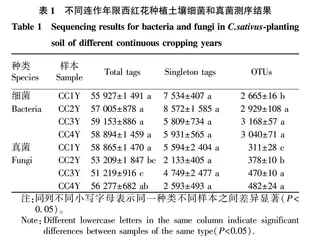
摘要 [目的]探究不同连作年限西红花种植土壤中细菌和真菌群落多样性以及网络结构特征。[方法]采用高通量测序技术分析不同连作年限西红花种植土壤微生物群落的多样性、组成以及网络特征差异。[结果]不同连作年限西红花种植土壤细菌和真菌的多样性发生明显变化,尤其是真菌的OTUs、Shannon、Simpson、Chao和ACE指数,随着连作年限增加呈现明显增加的趋势。主坐标分析(PCoA)和非度量多维尺度分析(NMDS)结果表明,不同连作年限西红花种植土壤中细菌和真菌群落组成均有明显差异。放线菌门(Actinobacteria)、变形菌门(Proteobacteria)、酸酐菌门(Acidobacteria)、绿弯菌门(Chloroflexi)和浮霉菌门(Planctomycetes)为4个不同连作年限西红花种植土壤中丰度前5位的细菌门。子囊菌门(Ascomycota)在4个连作年限中均为第一大真菌门(相对丰度>55%)。种植4年的土壤中放线菌门(Actinobacteria)和芽单胞菌门(Gemmatimonadetes)的丰度低于种植1年、2年和3年。产油菌属(Solicoccozyma)在连作4年西红花土壤中丰度显著高于其他年限(P<0.05)。细菌和真菌的共发生网络分析表明,不同连作年限西红花种植土壤细菌和真菌呈现不同的连接模式。[结论]该研究描绘了不同连作年限西红花种植土壤微生物多样性、组成以及网络结构的差异,从微生物角度探讨西红花连作障碍机制,为解决西红花的连作障碍提供参考。
关键词 西红花;连作年限;土壤微生物;多样性;网络特征
中图分类号 R 282.2 文献标识码 A 文章编号 0517-6611(2024)24-0137-08
doi:10.3969/j.issn.0517-6611.2024.24.031
开放科学(资源服务)标识码(OSID):
Differences of Microbial Diversity and Network Characteristics in Crocus sativus-Planting Soil of Different Continuous Cropping Years
LI Meng-zhi1, FENG Mao2, GAO Li1 et al
(1.Henan Key Laboratory of Zhang Zhongjing Formulae and Herbs for Immunoregulation,Nanyang Institute of Technology,Nanyang,Henan 473000; 2. Nanyang Institute of Technology, Nanyang,Henan 473000)
Abstract [Objective]To explore the diversity and network structure of bacterial and fungal communities in soil microorganism of Crocus sativus in different years of continuous cropping.[Method]High-throughput sequencing was carried out to determine the diversity, composition and network structure of bacterial and fungal communities in soil microorganism of C. sativus. [Result]The diversity of bacteria and fungi in soil of C. sativus had significant changes, especially the OTUs, Shannon,Simpson, Chao and ACE indices of fungi, which showed a significant increase trend with the increase of continuous cropping years. The results of PCoA and NMDS showed that there were significant differences in the composition of bacteria and fungi in soil of C. sativus in different years of continuous cropping.At the bacterial phylum level, Actinobacteria, Proteobacteria, Acidobacteria, Chloroflexi and Planctomycetes were the top five phyla.At the fungal phylum level, Ascomycota (55%) was the most abundant phylum. The relative abundance of Actinobacteria and Gemmatimonadetes were lower in soils of continuous cropping 4 year than continuous cropping 1,2 and 3 year. The relative abundance of Solicoccozyma was signifcantly higher in soils of continuous cropping 4 year than continuous cropping 1, 2 and 3 year (P<0.05). There were different connectivity patterns of bacteria and fungi in C. sativus-planting soil of different continuous cropping years.[Conclusion]The study describes the differences of microbial diversity, composition and network structure in C. sativus-planting soil of different continuous cropping years,and explores the mechanism of C. sativus continuous cropping obstacles from a microbial perspective, providing reference for solving the obstacles of continuous cropping in C. sativus.
Key words Crocus sativus;Continuous cropping years;Soil microorganism;Diversity;Network characteristic
西红花为鸢尾科植物番红花(Crocus sativus L.)的干燥柱头[1]。西红花主要在伊朗、西班牙、摩洛哥、土耳其、印度、希腊、意大利等地种植,目前在我国主要产地为上海、浙江、安徽和河南等省(市)[2-3]。相关的研究表明,西红花的柱头具有较多的药理作用,例如抗氧化、抗炎、抗抑郁、保护神经、保肝等,这些疗效主要来源于藏红花素、藏红花苷和藏红花醛3个主要活性成分[4]。由于西红花的化学成分具有重要的药理作用,导致西红花的重要性和需求显著增加[5]。然而,由于各种因素的影响,近几十年来世界范围内西红花的生产和种植呈下降趋势[6]。西红花的产量可能受到低温、干旱、水涝以及病原菌的影响[2]。
西红花为多年生药用植物,连续种植使病虫害加重,导致西红花的产量和品质下降,连作障碍明显[2]。土壤微生物在土壤功能中扮演重要角色,尤其是在调节植物生长、产量、品质等方面[7]。土壤中微生物的数量、种类、多样性以及群落结构对于维持土壤系统稳定和调节土壤生态系统功能至关重要[8]。前人很多的研究结果证实,连作打破了土壤中微生物的群落结构,作为药用植物三七(Panax notoginseng)[9]、人参(Panax ginseng)[10]、地黄(Rehmannia glutinosa)[11]等产生连作障碍的一个重要原因。连作改变土壤微生物多样性和组成,对土壤生产力和作物产量产生负面影响[12]。然而,由于西红花在我国种植历史不长,目前鲜见西红花不同连作年限土壤微生物多样性、群落组成的研究。因此,全面解析不同连作年限西红花土壤微生物多样性、群落组成,对于解析西红花连作障碍发生机制及预防其发生具有重要意义。该研究通过高通量测序获得不同连作年限西红花土壤微生物群落多样性和组成,阐明不同连作年限西红花种植土壤微生物的多样性、组成以及网络结构的差异,以期为解决西红花连作障碍提供依据。
1 材料与方法
1.1 样品采集
2021年10月20日(西红花花期)在河南省南阳市卧龙区蒲山镇马营村西红花种植基地采集土壤样本。分别采集连续种植西红花1年、2年、3年和4年的耕作层土壤,分别标记为CC1Y、CC2Y、CC3Y、CC4Y,每个种植年限选取3个种植小区,小区面积为4.0 m×1.5 m=6 m2。采取五点取样法用土钻采集深度为0~20 cm的耕层土壤放入无菌袋中,将5个点的土壤样品混在一起,作为一个生物学重复,共获得12个土壤样本。将土壤样本置于冰上运送到实验室,-80 ℃保存备用。
1.2 DNA提取与PCR扩增
取0.5 g土壤样品,按照试剂盒(Fast DNA TM Spin kit for soil)操作流程进行,获得土壤总DNA,用Nanodrop 2000对DNA样本的浓度和纯度进行检测;使用1%的琼脂糖凝胶对DNA质量进行检测,检测合格的NDA用于后续的PCR扩增。细菌16S的V3~V4区进行扩增,引物序列为341F(5′-CCTACGGGNGGCWGCAG-3′)和806R(5′-GGACTACHVGGGTATCTAAT -3′)[13];真菌ITS的ITS2区进行扩增,引物序列为ITS3_KYO2(5′-GATGAAGAACGYAGYRAA-3′)和ITS4(5′-TCCTCCGCTTATTGATATGC -3′)[14]。将上述引物分别合成带有barcode标签的特异性引物,进行PCR扩增,PCR扩增条件参考文献[15]进行。将纯化后的扩增产物(扩增子)连接测序接头,构建测序文库,在广州基迪奥生物科技有限公司的illumina Hiseq TM2500平台上机测序。
1.3 生物信息学分析
测序得到Raw reads,运用FASTP(version 0.18.0)过滤低质量的reads,利用FLSAH(version 1.2.11)将双端reads拼接为tag。为了获得高质量的clean tags,通过过滤条件将 raw tags 中的噪声序列过滤,然后运用UPARSE(version 9.2.64)系统将相似度≥97%的clean tags 聚类成OTUs[16]。运用UCHIME算法将所有嵌合体的tags去除[17]。16S和ITS的代表性序列集合使用RDP Classifier 的Nave Bayesian assignment 算法分别依据SILVA 和ITS2数据库进行物种注释(设定置信度的阈值为0.8)[18-19]。多样性指数通过QIIME软件(http://qiime.org/index.html)计算。基于Bray-unifrac距离的主坐标分析(principal coordinates analysis,PCoA)和基于Weighted-unifrac距离的非度量多维尺度分析(non-metric multidimensional scaling ordination,NMDS),通过R语言作图。运用Linear discriminant analysis effect size(LEfSe,v1.9.0)筛选西红花不同种植年限土壤中差异的微生物菌群[20]。“共发生”分析通过Python中的“SparCC”模块实现,网络图的可视化和属性测量利用交互平台Gephi进行。