鮰爱德华菌密度感应系统LuxI/LuxR的克隆及生物信息学分析
作者: 郭锦志 马士杰 韩先干 付立霞
摘要 克隆鮰爱德华菌(Edwardsiella ictaluri)YCH株酰基高丝氨酸内酯(Acyl homoserine lactones,AHLs)合成酶(LuxI)和AHLs受体蛋白(LuxR)的全长基因,并对其进行生物信息学分析。序列分析结果显示,LuxI全长666 bp,共编码221个氨基酸;LuxR全长720 bp,编码239个氨基酸。构建LuxI/LuxR的系统发生树,结果显示,鮰爱德华菌LuxI/LuxR与迟缓爱德华菌(Edwardsiella tarda)、杀鱼爱德华菌(Edwardsiella piscicida)、保科爱德华菌(Edwardsiella hoshinae)等有较近亲缘关系。利用SWISS-Model软件,分别以1K4J.1.A(46.15%)和5l09.1.A(40.83%)蛋白作为模板对LuxI蛋白的8-189区域和LuxR蛋白2个功能结构域Pfam:Autoind_bind(9~151氨基酸)和HTH_LUXR(171~228氨基酸)进行分子模拟,其中LuxI结构域内有明显的“V”形疏水腔结构,LuxR的N-端结构域有α-β-α排列,C-端结构域内有螺旋—转角—螺旋结构。
关键词 鮰爱德华菌;密度感应系统;LuxI/LuxR;基因克隆;生物信息学
中图分类号 S 941.42文献标识码 A文章编号 0517-6611(2023)18-0107-08
doi:10.3969/j.issn.0517-6611.2023.18.024
Molecular Cloning and Bioinformatics Analysis of Quorum Sensing System LuxI/LuxR of Edwardsiella ictaluri
GUO Jin-zhi1,MA Shi-jie1,HAN Xian-gan2 et al
(1. Yangzhou University College of Animal Science and Technology,Yangzhou, Jiangsu 225009;2. Shanghai Veterinary Research Institute,Chinese Academy of Agricultural Sciences,Shanghai 200241)
Abstract The full-length genes of Acyl homoserine lactones (AHLs) synthase (LuxI) and AHLs receptor protein (LuxR) of YCH strain of Edwardsiella ictaluri were cloned and bioinformatics was performed. Sequence analysis showed that LuxI was 666 bp in length and encoded 221 amino acids. LuxR is 720 bp in length and encodes 239 amino acids. The phylogenetic tree of LuxI/LuxR was constructed, and the results showed that LuxI/LuxR of Edwardsiella ictaluri is closely related to Edwardsiella tarda, Edwardsiella piscicida, Edwardsiella hoshinae,etc. Using SWISS-Model software, 1K4J.1.A (46.15%) and 5L09.1.A (40.83%) were used as templates for molecular simulation of LuxI protein 8-189 region and LuxR protein Pfam:Autoind_bind (9-151 amino acid) and HTH_LUXR (171-228 amino acid). The LuxI functional domain has obvious V-shaped hydrophobic cavity structure, the N-terminal functional domain of LuxR has α-β-α arrangement, and the C-terminal functional domain has helix-rotation-helix structure. This paper lays a foundation for further research on the LuxI/LuxR-mediated, quorum sensing system of E.ictaluri, and also provides a reference for the prevention and treatment of diseases caused by it.
Key words Edwardsiella ictaluri;Quorum sensing system;LuxI/LuxR;Gene cloning;Bioinformatic
鮰爱德华菌(Edwardsiella ictaluri)在分类上隶属于肠杆菌科(Enterobacteriaceae)爱德华菌属(Edwardsiella),是导致斑点叉尾鮰肠型败血症的一种革兰阴性病原菌[1],近年来发现其亦可感染其他养殖鱼类,如黄颡鱼、罗非鱼、尖吻鲈和斑马鱼等[2-5],呈世界性流行,给养殖生产带来了巨大危害。鮰爱德华菌的致病性通常认为,与其产生的各类毒力因子密切相关,现今已报道的毒力因子有尿酶、O多糖、溶血素、III型和VI型分泌系统等[6-10]。尽管如此,当前对鮰爱德华菌的感染与致病机制仍不够明晰。
密度感应系统是普遍存在于细菌中的一种胞间信号传导系统,该系统依赖于细菌产生的一种称为自体诱导物(Autoinducer,AI)的信号分子。在革兰阴性菌中,介导密度感应的信号分子主要有2类,一类为酰基高丝氨酸内酯(Acyl homoserine lactones,AHLs)类化合物,另一类为脂类化合物,又称AI-2,前者主要参与细菌种内的信息交流,后者则主要介导细菌种间的信息交流[11]。细菌通过信号分子的浓度感知外周细菌的数量,进而调控相关基因的表达以响应细菌群体的密度变化。借助于这一机制,密度感应系统在细菌的生长、运动、感染和耐药等方面发挥着重要作用[12-13]。
与上述2类信号分子合成相关的基因分别为LuxI和LuxS,其中LuxI编码AHLs合成酶,而LuxS则为AI-2生物合成中关键因子S-核糖基高半胱氨酸酶的编码基因[14]。在已发布的鮰爱德华菌基因组中亦显示存在LuxI和LuxS同源序列,并对LuxS的功能进行了研究[15-16],但这些研究中的菌株均分离自斑点叉尾鮰,而已有研究显示不同来源和地区的鮰爱德华菌特性存在差异[17]。除此之外,迄今尚鲜见鮰爱德华菌LuxI及其转录调控因子LuxR介导的密度感应系统的报道。鉴于此,笔者通过对1株黄颡鱼源的鮰爱德华菌的LuxI/LuxR进行克隆,并运用生物信息学手段对其推导的氨基酸序列进行结构分析,以期为进一步研究鮰爱德华菌LuxI/LuxR介导的密度感应系统及鮰爱德华菌所致疾病的防治提供参考。
1 材料与方法
1.1 材料 鮰爱德华菌YCH,分离自患红头病黄颡鱼,由实验室保存;大肠杆菌DH5α感受态细胞购自南京诺唯赞生物科技有限公司。pMD19 T Vector,PrimeSTAR HS(Premix),DNA A-Tailing Kit,DL 2 000 DNA Marker等分子生物学试剂均为宝生物工程(大连)有限公司产品。基因组提取试剂盒DNA Mini Kit及胶回收试剂盒Gel/PCR DNA Fragments Kit等为Geneaid产品。脑心浸液培养基(BHI)、LB肉汤和LB琼脂等培养基购自北京陆桥技术股份有限公司。鮰爱德华菌日常培养于BHI,大肠杆菌日常培养于LB肉汤,培养温度分别为28 ℃和37 ℃。
1.2 试验方法
1.2.1 引物设计与合成。
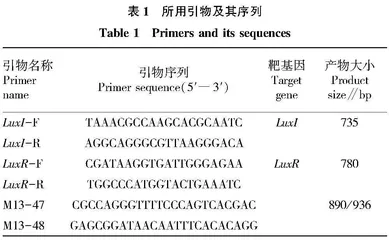
根据网上已发布的鮰爱德华菌菌株93-146基因组序列(NC_012779.2),利用Vector NTI 11.5.3 软件进行引物设计。考虑到不同来源菌株序列可能存在细微差异,为保证克隆基因测序结果的准确性,引物设计靶序列延展至相应基因的上、下游25~46 bp区域,以避免仅根据发布基因5′和3′进行引物设计可能对待克隆基因造成的潜在突变。所有引物(表1)均由赛默飞世尔(Thermo Fisher)旗下英潍捷基(上海)贸易有限公司合成。
1.2.2 细菌基因组提取。
将30%甘油冷冻保存的鮰爱德华菌接种至BHI液体培养基中复苏,然后以1∶50的比例传代接种,置于28 ℃气浴恒温摇床中以200 r/min转速振荡培养过夜,次日取3 mL菌液分装于1.5 mL微量离心管中,以12 000 r/min转速离心3 min,然后并管收集菌体,利用试剂盒DNA Mini Kit提取鮰爱德华菌基因组,相关操作按照产品说明书进行,制备的基因组DNA于20 ℃条件下保存备用。
1.2.3 LuxI和LuxR基因的PCR扩增。
以提取的鮰爱德华菌YCH基因组为模板,利用引物LuxI-F/LuxI-R和LuxR-F/LuxR-R分别对基因LuxI和LuxR进行PCR扩增,反应体系为50 μL,其中模板和正、反向引物各1 μL,PrimeSTAR HS(Premix)25 μL,最后用灭菌双蒸水补足体积。PCR反应程序:98 ℃ 30 s,55 ℃ 30 s,72 ℃ 30 s,30个循环,最后72 ℃延伸2 min。PCR产物经1% 琼脂糖凝胶电泳后,在凝胶成像系统中进行观察拍照。
1.2.4 目的片段的连接和测序。
在紫外分析仪中切下目的片段,通过胶回收试剂盒Gel/PCR DNA Fragments Kit回收目的片段,然后利用DNA A-Tailing Kit对回收片段的平滑末端进行加“A”操作,再与pMD19 T载体进行连接。连接产物通过热激转化大肠杆菌DH5α感受态细胞,然后均匀涂布于含氨苄青霉素(100μg/mL)的LB抗性平板,放入培养箱中于37 ℃过夜培养。待平板上长出菌落后,用灭菌牙签挑取单个菌落,接种于含氨苄青霉素的LB液体培养基中振荡培养,利用引物M13-47/M13-48进行PCR鉴定,PCR鉴定为阳性的克隆外送上海生物工程有限公司测序。
1.2.5 生物信息学分析。
将获得序列通过NCBI的BLAST(https://blast.ncbi.nlm.nih.gov/Blast.cgi)进行同源性分析,并从中选取部分序列利用MEGA-11软件构建系统进化树。利用丹麦技术大学生物信息学平台(http://www.cbs.dtu.dk/services/)SignalP、TMHMM和NetPhosBac分别进行信号肽、跨膜结构域和磷酸化位点预测。利用瑞士生物信息学研究所ExPASy平台(https://www.expasy.org/)Translate推导氨基酸序列,ProtParam确定开放阅读框(ORF)并预测分子质量和理论等电点(pI)等理化特性,SWISS-MODEL构建3D结构。利用ProtScale(https://web.expasy.org/protscale/)分析蛋白氨基酸序列的亲/疏水性。用PSORTb(https://www.psort.org/psortb/)预测亚细胞定位,SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/secpred_sopma.pl)预测蛋白质二级结构,SMART程序(http://smart.embl.de/)预测结构功能域。