基于trnL-trnF基因序列鉴别魔芋属核心种质资源
作者: 王孟 杨朝柱 覃宇 赵青华
摘要 采用株芽魔芋(A.mueller)、甜魔芋(A.paeoniifolius‘Yellow’)、滇魔芋(A.yunnesis)、勐海魔芋(A.kachinensis)、攸乐魔芋(A.yuleonsis)、梳毛魔芋(A.sinensis)、西盟(A.krausei)、A.asterostigmatus、白魔芋(A.albus)、花魔芋(A.konjac)、杂交魔芋等24个魔芋属植物作为鉴别的种内、种间植物。利用叶绿体基因trnL-trnF对样本DNA进行PCR扩增,采用双向测序,测序结果使用BioEdit进行校对,其序列在NCBI比对,最后利用MEGA 8.0构建系统发育树。结果表明:24种魔芋属的DNA扩增效率为100%,在NCBI的最高一致性在96.31%~100%,均属于魔芋属。表明trnL-trnF引物通用性好,特异性高,其引物可用于鉴别物种,分析其亲缘关系,为研究魔芋杂交育种中亲本选配提供参考。
关键词 魔芋;trnL-trnF;系统进化树
中图分类号 S 632.3 文献标识码 A 文章编号 0517-6611(2025)05-0093-03
doi:10.3969/j.issn.0517-6611.2025.05.018
Identification of Core Germplasm Resources of Amorphophallus Based on trnL-trnF Gene Sequence
WANG Meng 3, YANG Chao-zhu 3, QIN Yu 3 et al
(1.Enshi Tujia &Miao Autonomous Prefecture Academy of Agricultural Sciences, Enshi, Hubei 445000;2.Engineering Technology Center of Konjac Seed & Seedlings in Hubei Province, Enshi, Hubei 445000;3.Western Hubei Comprehensive Experimental Station, Hubei Agricultural Science & Technology Innovation Center, Enshi, Hubei 445000)
Abstract 24 Amorphophallus plants such as A.mueller, A.pmuelleraeoniifolius‘Yellow’, A.yunnesis, A.kachinensis, A.yuleonsis, A.sinensis, A.krausei, A.asterostigmatus, A. albus, A. konjac, hybrid konjac were used as intraspecific and interspecific plants for identification.The extracted DNA was amplified by PCR with chloroplast gene trnL-trnF, and sequenced in the first generation of the two-terminal sequencing strategy.The sequencing results were compared and analyzed with BioEdit, and the sequences were compared on NCBI. Finally, the phylogenetic tree was constructed with MEGA 8.0. The results showed that the DNA amplification efficiency of 24 species of Amorphophallus was 100%, and the best identity in NCBI was 96.31%-100%,all belong to Amorphophallus. The results showed that the primers of trnL-trnF had good universality and high specificity, and the primers could be used to quickly identify species and analyze their genetic relationship, providing reference for the study of parental selection in konjac cross breeding.
Key words Konjac;trnL-trnF;Phylogenetic tree
魔芋属(Amorphophallus)特色经济作物,其资源丰富,主要分布在亚洲中南半岛和我国秦岭以南及东南亚和南亚印度等地。在国内有26种[1],主要分布在海拔800~1 200 m的山地。魔芋分布广,其资源的遗传多样性丰富[2-3],区分魔芋亲缘关系不仅对魔芋杂交育种有着重要作用,还可以避免魔芋产业发展的原材料使用出现混乱。但目前魔芋属的分类主要是通过形态学区别,形态学区分对魔芋基础研究要求较高,且易受外界环境的影响导致结果不精准。DNA条形码(DAN barcode)是物种鉴定和研究种间进化关系可靠的手段,其结果可靠,被广泛用于物种鉴别。
目前主要用的序列有ITS1、rbcL、matK、trnL-trnF、trnH-psbA。trnL-trnF序列是cp DNA的非编码区,其进化速率快,片段两端存在保守序列,是易于设计,通用性好,特异性高等特点的引物,叶绿体基因trnL-trnF已在甘薯[4]、草莓[5]、枸杞[6]等作物的分子鉴定、亲缘关系分析中广泛应用。Claudel等[7]利用ITS1、rbcL 、matK对157种魔芋属植物进行了分子系统发育分析。Zhao等[8]研究显示,trnH-psbA能有效鉴别3种魔芋属,但trnL-trnF的引物通用性比trnH-psbA低。笔者拟通过叶绿体trnl-trnH序列鉴别魔芋属材料,分析其亲缘关系,旨在为魔芋种质资源鉴别提供参考。
1 材料与方法
1.1 试验材料
供试魔芋材料:株芽魔芋(A.mueller)、甜魔芋(A.paeoniifolius‘Yellow’)、滇魔芋(A.yunnesis)、勐海魔芋(A.kachinensis)、攸乐魔芋(A.yuleonsis)、梳毛魔芋(A.sinensis)、西盟(A.krausei)、A.asterostigmatus、白魔芋(A.albus)、花魔芋(A.konjac)、鄂魔芋1号(YuanZa No.1)、2018H-66*2018B-4、2014-38*Y121-242、2014-106*BD164、Y1H81*A.corrugatus、2022H-34*2022B-21。
试验样品均取自恩施土家族苗族自治区农业科学院魔芋所种植圃,取健康、无病虫害新鲜叶片,采集消毒后做好标记,置于-80 ℃超低温冰箱保存备用。
1.2 试验方法
1.2.1 DNA提取方法。采用植物基因组DNA提取试剂盒 Plant Genomic DNA kit(天根生化科技有限公司,北京)改良后的方法[9]提取叶片DNA,植物样品从冰箱取出后,迅速用乙醇双面擦拭,取100 mg新鲜叶片置于2.0 mL离心管中,放入液氮罐装入液氮,迅速用铁质圆柱头类研磨,后续按照试剂盒的方法提取样本DNA。
1.2.2 引物设计。根据已知植物trnL-trnF序列,利用其保守位点和变异位点,用Primer Premier 5 软件设计,委托武汉生工合成部合成引物,合成引物按要求扩大10倍缓冲液。
1.2.3 PCR扩增及电泳。
PCR反应体系40 μL,包括13.8 μL ddH2O,3.0 μL Template,20.0 μL 2×Taq PCR Master Mix(采购于TIANGEN公司),3.2 μL Prinmer。PCR产物采用第一代双端测序,测序在武汉生工测序部完成。使用1.0%琼脂糖凝胶液静置30 min,倒入电泳槽中,用移液枪吸取PCR扩增产物4.0 μL,设置电泳(110 V),约50 min后取出并放置于凝胶成像系统中进行分析,比较后无杂带且清晰时拍照保存图像。
试验过程中所使用的仪器有恒温水浴锅(HH-4,坛市科信仪器厂),电泳仪(DYY-10C,北京市六一仪器厂),PCR仪(PCR System9700,Gene Company Limited基因有限公司),冷冻离心机(5417R,德国艾本德公司),凝胶成像系统(Gel Doc200,伯乐公司)。
1.2.4 数据分析。
利用BioEdit对测序获得的原始序列进行检查、拼接及校对,并将获得的序列在NCBI Gen Bank 数据库进行对比。比对后的序列使用MEGA 8.0软件进行序列比对,将处理后的数据导入 MEGA-X软件,进行 ClustalW对比,以邻接法(Neighbor-joining,NJ)构建系统发育树。扩增引物与扩增程序:碱基序列为5′-GGGCAATCCTGAGCCAAATC-3′,3′-GGGACTTGAAACCTCACGAT-5′;反应体系为40 μL,13.8 μL ddH2O,3.0 μL模板,20.0 μL Mix,1.6 μL引物;反应条件为94 ℃ 4 min,94 ℃ 45 s, 55 ℃ 45 s,72 ℃ 5 min,35个循环。
2 结果与分析
2.1 PCR 扩增
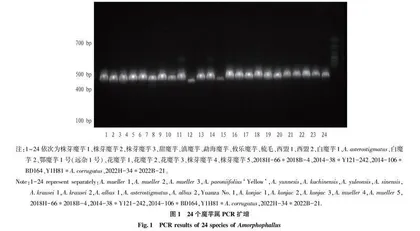
如图1所示,采用trnL-trnF(长度510 bp)在 魔芋样本中均获得了预期的扩增条带,扩增成功率为 100%。24个样本中包含株芽魔芋、甜魔芋、滇魔芋、勐海魔芋、攸乐魔芋、梳毛魔芋、西盟魔芋、白魔芋、A.asterostigmatus、鄂魔芋1号、花魔芋、田阳魔芋12个魔芋种质资源。
2.2 序列验证
PCR产物进行双向测序,测序成功率为100%,测序后序列通过BioEdit和DNAMEN 8进行校对,获
得24条序列,采 用 NCBI(National Center for Biotechnology Information)平台的BLAST 工具与数据库进行物种最佳匹配度匹配。比对结果见表1,比对结果显示,24条序列均属于魔芋属,A.konjac、2014-38*Y121-242、2014-106*BD164、YuanZa No.1最高一致性均为100%,其他材料最高一致性均在99.31%及以上,结果表明所使用引物是特异性高的有效扩增引物(表1)。
2.3 亲缘关系
24份魔芋属材料亲缘关系图2所示,杂交魔芋和花魔芋、白魔芋的亲缘关系较近,Y1H81*A corrugatus的特性更偏向白魔芋;A.asterostigmatus是由国外引进,至今未命名的魔芋种,其亲缘关系和西盟魔芋较近;西盟魔芋同花魔芋、白魔芋的亲缘关系近,其特性相似;滇魔芋、攸乐魔芋、勐海魔芋与梳毛魔芋关系较近;株芽魔芋、甜魔芋的亲缘关系差距较大,这可能是株芽魔芋种间差距大造成的。
3 结论与讨论
魔芋在我国种质资源丰富,且分布较广。DNA条形码技术不受环境影响,能快速、准确地对物种进行识别,为研究物种的进化、遗传变异、系统发育以及生物多样性等提供理论依据,是魔芋属种质资源鉴定的有效技术。该研究利用叶绿体基因trnL-trnF,通过PCR、电泳成像、BLAST核苷酸序列比对分析手段,验证了trnL-trnF引物的有效性,建立24个魔芋属的DNA条形码。trnL-trnF引物序列在24个遗传背景较大的魔芋样本中均获得了预期的扩增条带,扩增成功率为 100%,双向测序矫正后成功获得24条魔芋属的DNA条形码。序列在NCBI比对后,最佳匹配度在96.31%~100%,表明trnL-trnF引物通用性强,特异性高,是鉴别魔芋属的有效引物。亲缘关系的研究可为魔芋属种质资源利用与创新提供依据,该研究中西盟魔芋和花魔芋、白魔芋亲缘关系较近,这与李梅[10]的研究结果一致;攸乐魔芋与滇魔芋、勐海魔芋亲缘关系较近,这与牛义等[11-13]研究结果一致。董坤等[14]利用ISSR分子标记对魔芋材料进行聚类分析,结果显示,勐海魔芋、珠芽魔芋、滇魔芋、甜魔芋各自聚为一类,但该研究中株芽魔芋的5个样品的亲缘关系较远,这可能由于种间差异较大造成。魔芋作为特色的园艺植物,品种间形态特征变化较小,DNA条形码技术鉴定品种十分重要,所做亲缘关系图可为魔芋杂交育种中亲本选配、定向配组提供理论参考。