云南原始地方种四路糯基因组中的MITE类转座子“124”
作者: 武晓阳 杨亚洲 隆文杰 陈丹 周国雁 杜娟 伍少云 蔡青 黄兴奇
摘要[目的]研究四路糯与普通玉米之间的亲缘关系。[方法]比较四路糯与B73基因组中“124”转座子的构成差异。[结果]在B73基因组中鉴定出13个“124”转座子序列和其插入位点信息;通过重测序得到约120 Gb的四路糯基因组序列,并从中鉴定出17个“124”转座子序列和其插入位点信息;在二者基因组中存在3个共同的“124”插入位点,这些位点展示出“124”转座子在序列和插入位点一致;B73基因组和四路糯基因组中均存在与waxy基因中“124”一致的转座子序列;建立的系统进化树可将“124”转座子归为4种类型,其中并没有单一基因组来源的类型。[结论]四路糯与B73基因组的差异是玉米种内的差异。
关键词糯玉米地方品种;四路糯;重测序;“124”转座子;起源演化
中图分类号S513文献标识码A
文章编号0517-6611(2022)13-0093-05
doi:10.3969/j.issn.0517-6611.2022.13.025
The MITE Transposon “124” in the Genome of Yunnan Original Landrace Four-Row Wax
WU Xiao-yang YANG Ya-zhou LONG Wen-jie et al(1. School of Life Sciences, Yunnan University, Kunming, Yunnan 650091;2. Biotechnology and Germplasm Resources Institute, Yunnan Academy of Agricultural Sciences, Kunming, Yunnan 650223;3. Life Science and Technology Center, China Seed Group Co., Ltd., Wuhan, Hubei 430206)
Abstract[Objective]To study the genetic relationship between Four-Row Wax and common maize.[Method]The differences in the composition of “124” transposon in the genomes of Four-Row Wax and B73 was compared.[Result]The results showed that 13 “124” transposon sequences and their insertion sites were obtained in B73 genome;about 120 GB Four-Row Wax genome sequence was obtained by resequencing, from which 17 “124” transposon sequences and their insertion sites were identified;there were three common sites in the two genomes, which showed the sequence and insertion sites consistency;there are “124” transposons in B73 and Four-Row Wax genomes, which are consistent with “124” in waxy gene;the phylogenetic tree established can classify “124” transposons into four types, among which there is no type from a single genome.[Conclusion]The results showed that the genomic differences between Four-Row Wax and B73 are intraspecific.
Key wordsWaxy Maize Landraces;Four-Row Wax;Resequencing;“124” transposon;Origin and evolution
糯玉米(Zea mays L.var.certaina Kulesh)是一类主要的鲜食玉米。糯玉米籽粒胚乳中的淀粉全部为支链淀粉,使得籽粒具有糯性口感。糯性受隐性的waxy单基因控制。普通玉米waxy基因能正常表达GBSSⅠ(Granule-Bound Starch Synthase Ⅰ)蛋白,用于直链淀粉的合成。waxy的突变造成GBSSⅠ蛋白失去活性,直链淀粉的合成受阻,使玉米籽粒中的淀粉几乎全为支链淀粉,产生出籽粒的糯性[1-2]。玉米waxy基因位于第9号染色体上,全长约4.5 kb,包含有14个外显子[3]。
糯玉米起源于我国,我国拥有丰富的糯玉米地方品种,大量的waxy等位变异也存在其中,主要waxy等位基因类型包括wx-D7、wx-D10、wx-Cin4、wx-124、wx-Reina、wx-xuanwei、wx-PIF/Harbinger、wx-hAT和wx-Elote2等类型。wx-D7、wx-D10是序列缺失造成的突变[4-6];wx-Cin4、wx-124、wx-Reina、wx-xuanwei、wx-PIF/Harbinger、wx-hAT和wx-Elote2是玉米基因组中转座子活动造成的突变[7-10]。其中wx-124是由MITE类转座子“124”插入waxy基因的第六外显子造成的突变[7]。
转座子插入是造成waxy基因突变的主要原因之一[11]。转座子是基因组中一段可移动的DNA序列,可以通过切割、重新整合等一系列过程从基因组的一个位置“跳跃”到另一个位置。根据转座子在基因组内跳跃媒介的不同,可以将转座子分为DNA转座子和RNA转座子,RNA转座子又叫反转录转座子[12]。RNA转座子通过“复制—粘贴”的方式转座,DNA转座子通过“剪切—粘贴”的方式转座。微型反向重复转座元件(miniature inverted-repeat transposable element,MITE)是一类主要的DNA转座子[13],TITE转座子通常具有靶位点重复(target site duplications,TSDs)和末端反向重复(Terminal Inverted Repeats,TIRs)的序列结构,根据这些特征其进一步可以划分成Tc1/Mariner、Mutator、hAT、CACTA、PIF/Harbinger、P和Novosib等家族类型[14]。“124”是在waxy基因突变研究中,鉴定出的一个新的MITE类转座子家族[7]。
云南及其周边地区是我国少数民族的聚集地,自古以来就有食用糯性食物的习惯。很多特殊类型的糯性玉米就是在这一地区发现[15]。“四路糯”就是其中之一,最早在云南勐海的傣族居民区发现,其果穗具有四路籽粒,形态具有许多玉米原始特征,即分蘖多、果穗多、穗行数少等,并具有独特的口感[16-17]。“勐海四路”玉米已经用于许多科学研究中。通过对waxy基因的鉴定,发现勐海四路是wx-D10类型的突变[4-5];通过“四路糯”玉米穗行数QTL的定位,鉴定出多个控制穗行数的主效QTL位点[18-20];“勐海四路”基因组重测序的完成,为“四路糯”的研究提供了基础数据[21]。由此可见,“四路糯”已成为玉米遗传育种及相关基础学科的特异研究材料,收集保护好这一珍贵的我国古老地方品种具有重要意义。云南省农业科学院作物种质资源库经过多年的考察收集,保存了云南不同地区的四路糯玉米资源[22]。笔者对来自中缅边境地区的四路糯进行了基因组的重测序,通过与普通玉米B73基因组中“124”转座子进行比较,探讨了四路糯与普通玉米之间的亲缘关系。
1材料与方法
1.1植物材料以糯玉米地方品种四路糯为研究材料,用于其基因组的重测序。试验材料由云南省农业科学院种质资源库收集保存。
1.2基因组重测序对四路糯中随机选取的3个单株分别进行了基因组的重测序。样品DNA提取参考文献[23],检验合格的DNA样品通过Covaris破碎机随机打断成长度为350 bp的片段。采用TruSeq Library Construction Kit 试剂盒进行建库。文库构建完成后,先使用Qubit 2.0进行初步定量,稀释文库至1 ng/μL,随后使用Agilent 2100对文库的insert size进行检测,insert size符合预期后,使用Q-PCR方法对文库的有效浓度进行准确定量,使文库有效浓度大于2 nmol/L,以保证文库质量。库检合格,根据文库的有效浓度及数据产出需求进行 illumina HiSeq 双向测序。获得原始测序序列(sequenced reads)后,进行接头序列及polyN,polyA 等序列的过滤,将过滤后的valid reads测序数据用于进一步分析。
1.3“124”在B73基因组中的鉴定使用玉米自交系B73的基因组序列(http://ftp.maizesequence.org/current/assembly/),建立本地Blast数据库[24]。使用wx-124中的“124”序列比对B73基因组,采用Blastn[25]程度确定“124”的插入位点,提取插入位点上下游150 bp的序列,用于分析“124”的结构。
1.4“124”在四路糯基因组中的鉴定根据“124”的结构特征编写Perl代码(图1),提取测序结果中具有“124”结构的Read序列;使用获得的Read序列建立本地Blast数据库;利用Blastn[25]程序进一步确定含有“124”转座子的Read序列;利用这些Read序列分析其中的“124”结构;根据“124”序列插入位点信息和剔除其中的重复。
1.5序列分析使用RNAstructure 6.2软件预测转座子“124”的二级结构[26];使用软件CLUSTAL W和Genedoc进行序列比对[27-28];使用MEGA 5[29]软件构建最大似然树(Maximum Likelihood Tree)。植物MITE数据库(Plant MITE Database,http:∥pmite.hzau.edu.cn/)用于 “124”转座子家族的划分[14]。
2结果与分析
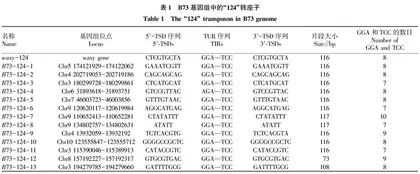
2.1B73基因组中“124”“124”家族的转座子最早在waxy基因中鉴定出。通过与数据库中已知的MITE比对,未发现相似转座子,由此可知“124”为新鉴定出的转座子家族。该研究使用这一序列与B73基因组进行比对,进一步鉴定出13个“124”家族转座子的插入位点,利用转座子上下游150 bp的序列进一步分析出每个转座子的结构,将这些序列命名为B73-124-1~ B73-124-13(表1)。“124”在玉米基因组中是一类低拷贝数的转座子家族。典型的“124”转座子全长116 bp;具有9 bp靶位点的重复序列(TSDs),但这一结构的序列并不保守;具有保守的3 bp末端反向互补重复序列(TIRs)5′-GGA-3′和5′-TCC-3′;转座子序列内部存在多处5′-GGA-3′和5′-TCC-3′序列,存在进一步截短的趋势,最短的“124”序列仅73 bp;“124”转座子可形成明显的二级结构(图2),“124”家族成员的序列存在一定的相似性,但相似程度不高。值得注意的是,wx-124中的“124”序列与B73-124-1的序列完全一致,但二者的靶位点TSDs结构并不相同。